monocle3算法
用途
利用monocle3 算法对空间转录组和单细胞数据进行轨迹推断,自动输出多种轨迹相关结果。
运行方式
- 使用细胞注释后的h5ad,准备rds文件
SDAS dataProcess h5ad2rds -i st.h5ad -o outdir
- 运行monocle3
SDAS trajectory monocle3 -i st.rds -o outdir --root_key anno_spotlight --root CAF_DES \
--gene_symbol_key real_gene_name \
--batch_key sampleID
输入参数说明
| 参数 | 是否必须 | 默认值 | 描述 |
|---|---|---|---|
| -i / --input | 是 | rds文件,要求有原始表达矩阵 | |
| -o / --output | 是 | 输出文件夹 | |
| --root_key | 是 | meta.data中根节点所在的列名称 | |
| --root | 是 | 设置为根节点的名称 | |
| --assay | 否 | rds文件中用于分析的assay名称,若不设置则用当前默认assay | |
| --gene_symbol_key | 否 | real_gene_name | rds文件中基因名(symbol)的列名(meta.features),_index 表示用矩阵的列名 |
| --batch_key | 否 | 进行批次校正的meta.data列名,不输入则不做去批次 | |
| --resolution | 否 | leiden聚类的resolution参数,不输入则算法会自适配 | |
| --use_existing_umap_cluster | 否 | 使用rds已有的umap和cluster信息 | |
| --umap_key | 否 | umap | 输入rds存储umap信息的名称 |
| --cluster_key | 否 | leiden | 输入rds存储cluster信息的名称 |
| --deg | 否 | 分析随拟时序变化的差异基因则输入”--deg” | |
| --n_cpus | 否 | 8 | 多线程数 |
| --top_gene_num | 否 | 5 | 随拟时序变化的top差异基因数目的表达图 |
| --gene_file | 否 | 自定义基因列表文件路径,画基因随拟时序变化的表达趋势图 | |
| --gene_color_label | 否 | pseudotime | 基因图展示的列的名称 |
| --pval_cutoff | 否 | 0.05 | 差异基因筛选p值 |
| --qval_cutoff | 否 | 0.05 | 差异基因筛选q值 |
| --seed | 否 | 42 | 随机种子 |
输出结果展示
| 结果文件 | 描述 |
|---|---|
<input_name>_dimension.png/pdf |
降维图,展示细胞在低维空间的分布结构 |
<input_name>_dimension_color_by_batch.png/pdf |
以批次信息展示的降维图(有做批次校正时输出),用于评估批次效应 |
<input_name>_cluster.png/pdf |
聚类图,展示细胞聚类分布情况 |
<input_name>_roots.png/pdf |
root图,显示轨迹分析的根节点位置 |
<input_name>_pseudotime.png/pdf |
拟时序图,展示细胞的拟时序分布 |
<input_name>_top_genes_in_pseudotime.png/pdf |
拟时序变化的top基因表达趋势图 |
<input_name>_custom_genes_in_pseudotime.png/pdf |
拟时序变化的自定义基因表达趋势图 |
<input_name>_monocle3.rds |
包含轨迹分析结果的rds文件 |
<input_name>_pseudotime.csv |
细胞的拟时序结果,记录每个细胞的拟时序值 |
<input_name>_deg_trajectory.xls |
随拟时序变化的所有基因的结果 |
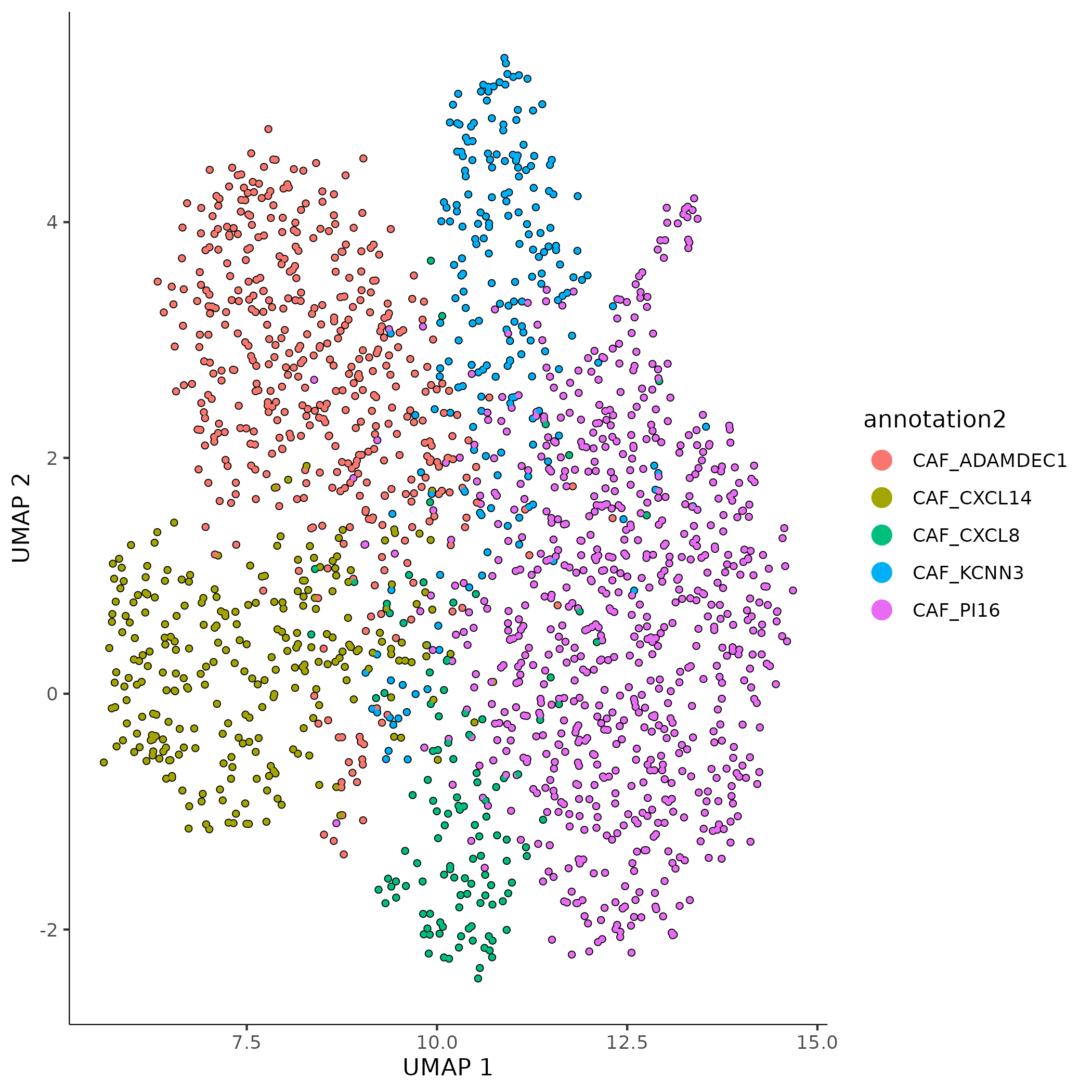
- 降维图:
<input_name>_dimension.png/pdf展示细胞在低维空间的分布结构,反映整体异质性和分群结构
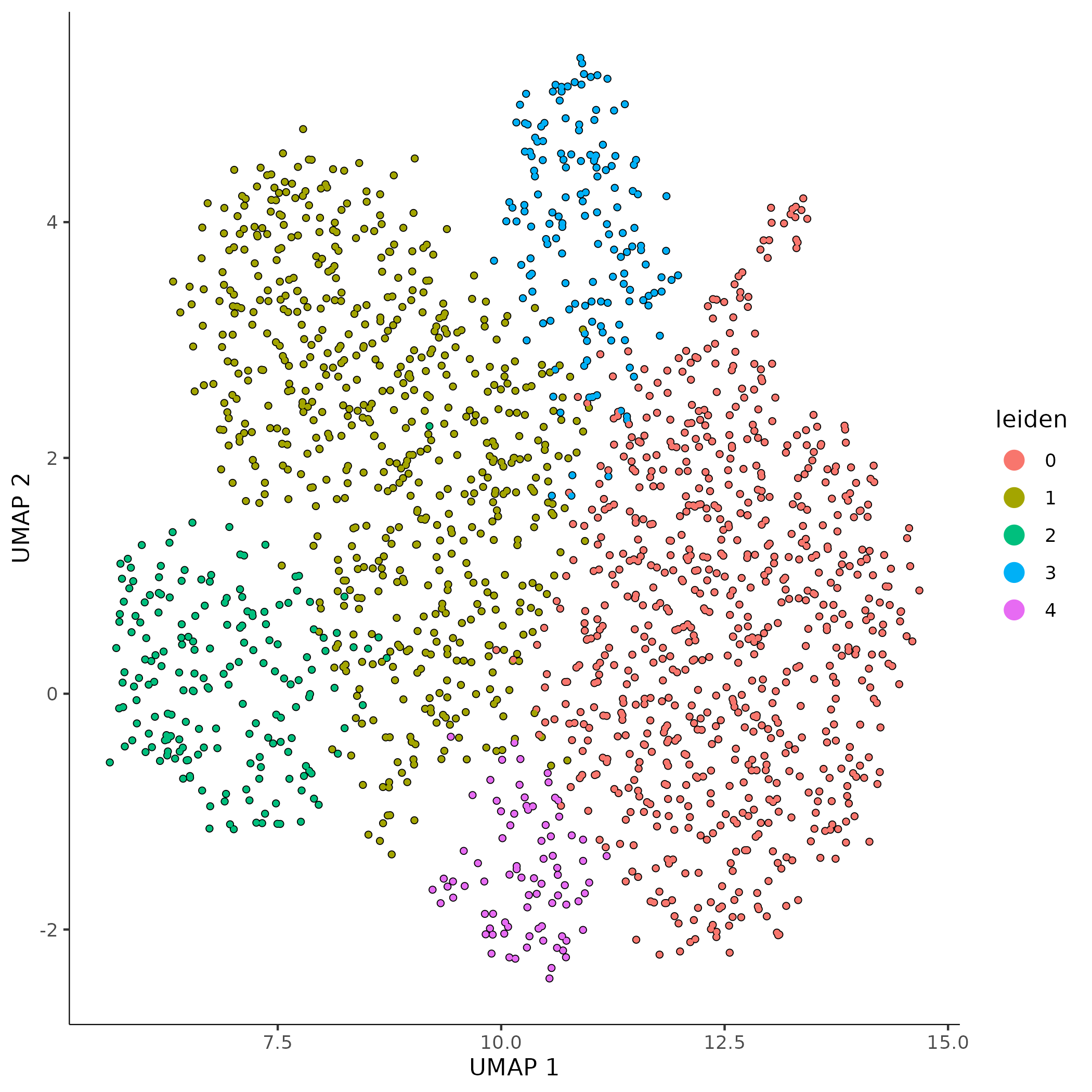
- 聚类图:
<input_name>_cluster.png/pdf展示细胞聚类分布,反映整体异质性和分群结构
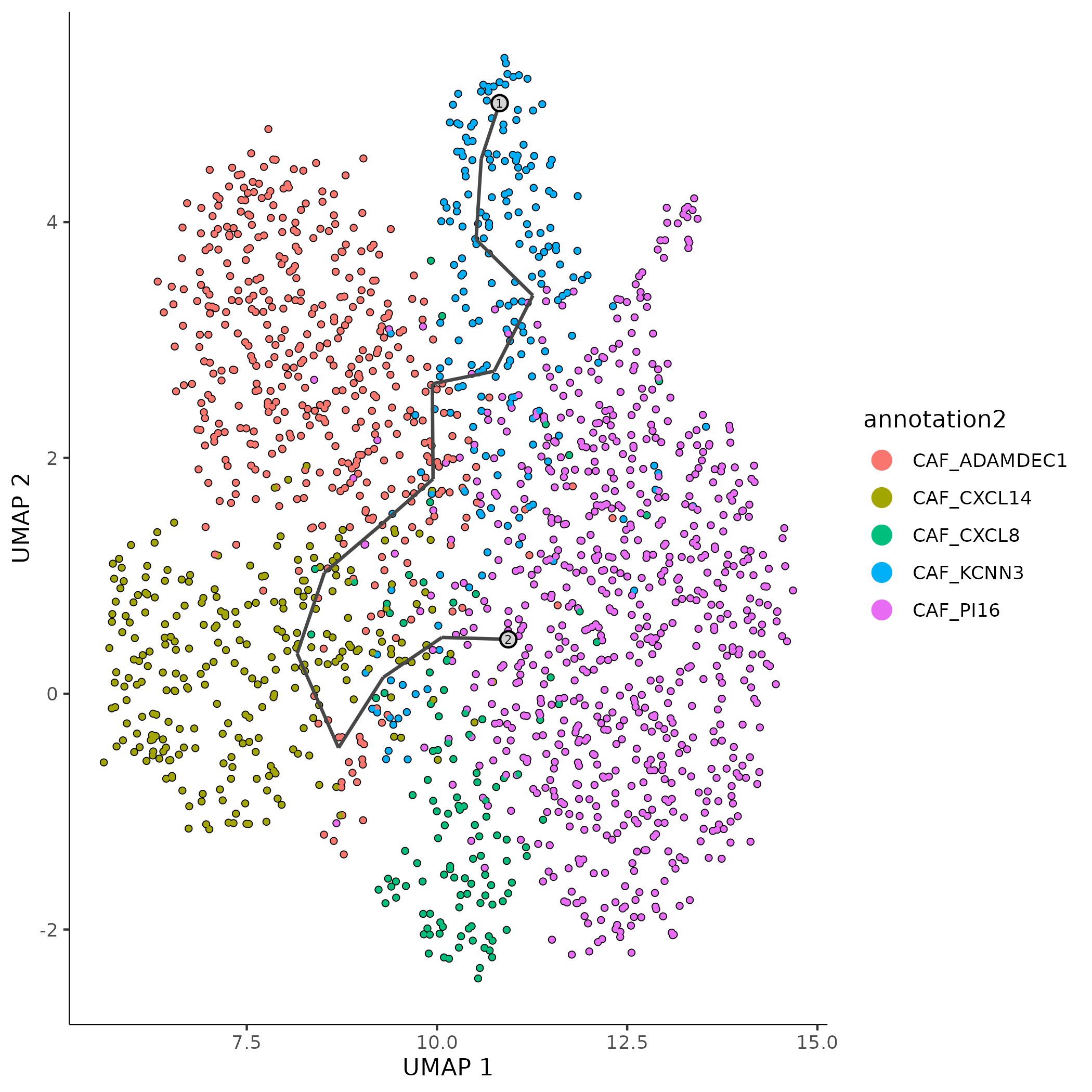
- root图:
<input_name>_roots.png/pdf展示轨迹分析的根节点,标记轨迹推断的起始点(根节点),用于后续拟时序排序
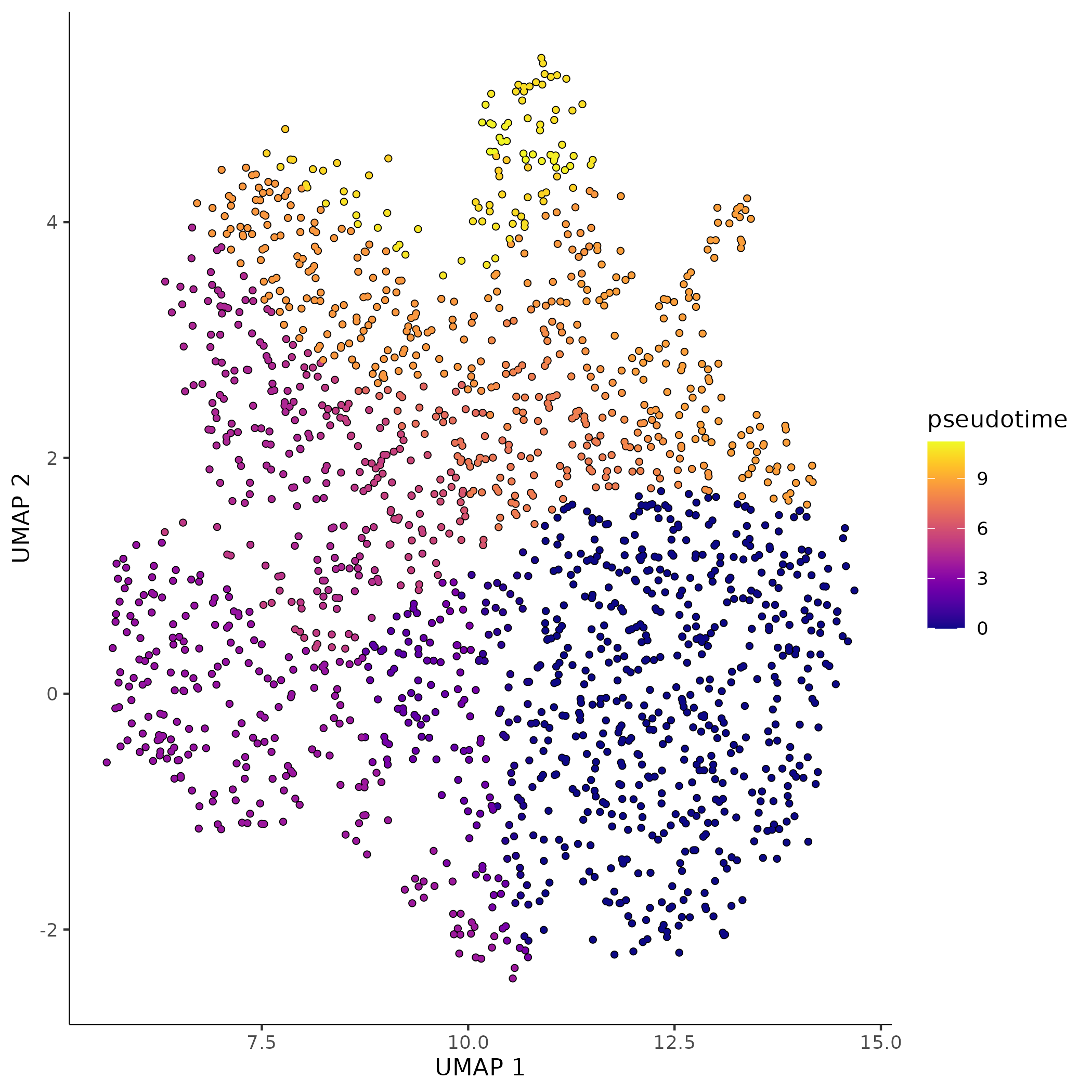
- 拟时序图:
<input_name>_pseudotime.png/pdf展示细胞拟时序分布,细胞按推断的发育/转化顺序着色,反映动态变化过程
- 拟时序变化的top基因表达趋势图:
<input_name>_top_genes_in_pseudotime.png/pdf展示拟时序变化最显著的top基因表达趋势
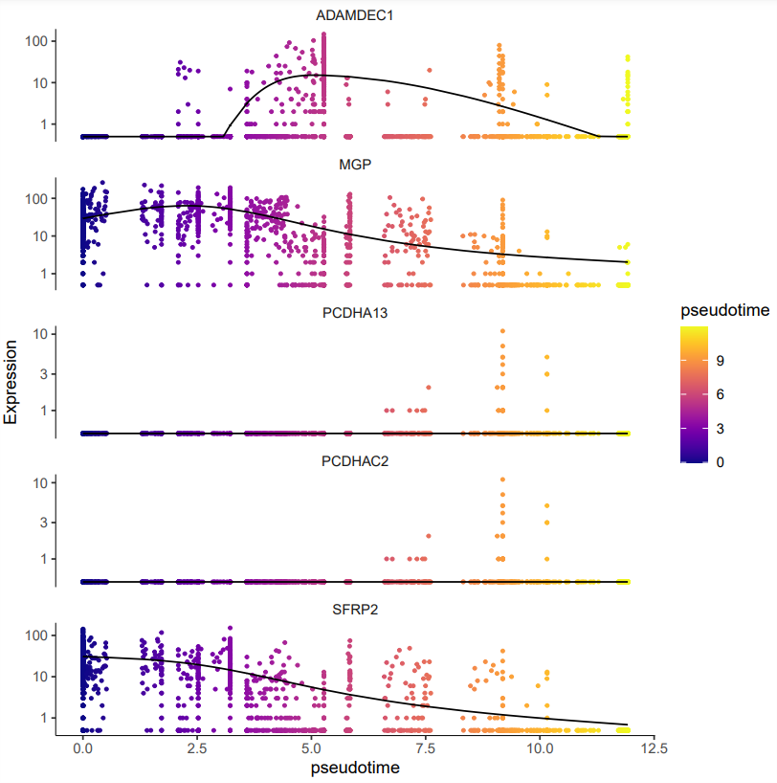
- 拟时序变化的自定义基因表达趋势图:
<input_name>_custom_genes_in_pseudotime.png/pdf展示用户指定基因在拟时序过程中的基因表达趋势

调参建议
monocle3的轨迹分析一般需以细胞亚型精准注释为前提。通过亚型划分,算法可排除无关细胞干扰,聚焦于具有发育连续性的细胞群体,从而推断符合生物学逻辑的分化路径。实际分析中,推荐选取特定细胞群体,不要使用全片进行分析,同时可降低运行时间和内存。