CRAWDAD算法
用途
利用 CRAWDAD (Correlated Analysis of WORkflow-Derived Spatial Associations and Distances) R包分析空间分辨转录组数据中细胞类型间的相互作用关系和空间共变趋势,识别显著的细胞邻近或回避现象。
运行方式
SDAS spatialRelate crawdad -i st.h5ad -o outdir --label_key anno_cell2location \
--spatial_coords_scale 0.5 \
--crawdad_scales_definition '100,1000,10' \
--crawdad_perms 10 \
--crawdad_neighborhood_dist 50 \
--n_cpus 24 \
--seed 42
输入参数说明
| CRAWDAD 参数 | 是否必须 | 默认值 | 描述 |
|---|---|---|---|
| -i / --input | 是 | 输入的 AnnData h5ad 文件路径。要求包含空间坐标和细胞注释。 | |
| -o / --output | 是 | 分析结果的输出文件夹路径。 | |
| --label_key | 是 | AnnData 对象中 (adata.obs) 用于细胞类型或区域聚类注释的列名。 | |
| --spatial_coords_scale | 否 | 0.5 | 应用于空间坐标的全局缩放因子。例如,对于stereo-seq,该值设为0.5,将坐标转为微米,方便结果解读 |
| --min_cells_per_type | 否 | 20 | 为纳入分析,每种细胞类型/聚类在筛选后所需的最少细胞(或 spot)数量。重要:建议设置为20及以上,过小的值可能导致分析不稳定。 |
| --crawdad_neighborhood_dist | 否 | 50 | CRAWDAD 分析中用于定义细胞邻域的距离(通常单位为微米)。影响 findTrends 函数中邻居细胞的识别。必须为正整数。重要:此参数应与细胞大小和空间分布特征匹配,建议设置为细胞直径的1-3倍。 |
| --crawdad_scales_definition | 否 | "100,1000,10" | CRAWDAD 分析的距离尺度定义,格式为 "起始距离,结束距离,点数"。例如 "50,500,10" 表示从距离50到500,均匀取10个距离点进行分析。起始距离必须非负,结束距离必须大于起始距离,点数必须为正整数。重要:起始距离应大于或等于crawdad_neighborhood_dist。 |
| --crawdad_perms | 否 | 10 | CRAWDAD R脚本中用于计算相互作用p值的置换次数。增加此值会提高p值的准确性但增加计算时间。必须为正整数。 |
| --n_cpus | 否 | 8 | 并行计算时可使用的 CPU核心数。CPU核数越多,运行时间相应减少,可根据计算资源合理设置。 |
| --seed | 否 | 42 | 用于可复现性的随机种子。必须为非负整数。 |
输出结果展示
| 结果文件 | 描述 |
|---|---|
<input_name>_crawdad_summary_coloc_dotplot.pdf/png |
汇总共定位点图:展示显著的细胞类型对共定位或互斥关系。点的大小/颜色代表强度/方向。视觉效果经过优化(点大小、X轴标签)。 |
<input_name>_crawdad_spatial_cluster_plot.pdf/png |
空间聚类图:按细胞类型 (label_key) 着色显示各类细胞的空间排布。点大小根据数据量动态调整。 |
<input_name>_crawdad_trend_plots_all.pdf |
互作趋势图 (多页PDF): 每页对应一个参考细胞类型,含空间分布子图 (点大小动态调整,保持长宽比) 和互作强度 (Z-score) 随距离变化子图。 |
<input_name>_crawdad_trend_plots_png/ (文件夹) |
与上述PDF每页内容相同的独立PNG图片。 |
<input_name>_crawdad_trend_plot_ref_<celltype_name>.png |
单个参考细胞类型的互作趋势组合图。 |
<input_name>_crawdad_melted_results.csv |
主要定量结果: 包含细胞类型间详细互作分析数据 (如参考细胞、邻居细胞、距离尺度、Z-score)。 |
<input_name>_crawdad_zsig.csv |
Bonferroni 校正后的Z-score显著性阈值 (zsig)。 |
<input_name>_crawdad_r_stdout.log |
R脚本标准输出日志 (INFO级别,带时间戳)。 |
<input_name>_crawdad_r_stderr.log |
R脚本标准错误日志 (WARNING/ERROR级别,带时间戳,含错误详情)。 |
(<input_name>_processed_for_crawdad.h5ad) |
(中间文件) 预处理后传递给R脚本的AnnData文件。 |
- 主要定量结果表格:
<input_name>_crawdad_melted_results.csvCRAWDAD输出结果展示细胞类型间详细互作分析数据,包含参考细胞、邻居细胞、距离尺度、Z-score等关键互作指标
| perm | neighbor | Z | scale | reference |
|---|---|---|---|---|
| 1 | B | 1.05670532268942 | 100 | B |
| 2 | B | -1.45780531937283 | 100 | B |
| 3 | B | -2.7310679139005 | 100 | B |
| 4 | B | -1.76600222336501 | 100 | B |
| 5 | B | -0.122214856981382 | 100 | B |
| 6 | B | -0.215950198738648 | 100 | B |
| 7 | B | -1.34972501073846 | 100 | B |
| 8 | B | -1.38061633431226 | 100 | B |
| 9 | B | -1.56577575976306 | 100 | B |
| 10 | B | -1.71982948316056 | 100 | B |
| 1 | Endo | 0.586389142807096 | 100 | B |
| 2 | Endo | 0.953148492138181 | 100 | B |
| 3 | Endo | 1.18876625358954 | 100 | B |
| 4 | Endo | 0.71926735855275 | 100 | B |
| 5 | Endo | 0.752573768964804 | 100 | B |
| 6 | Endo | 1.32419648335896 | 100 | B |
| 7 | Endo | 1.46020965216509 | 100 | B |
| 8 | Endo | 0.819291421388121 | 100 | B |
| 9 | Endo | 1.87180663929578 | 100 | B |
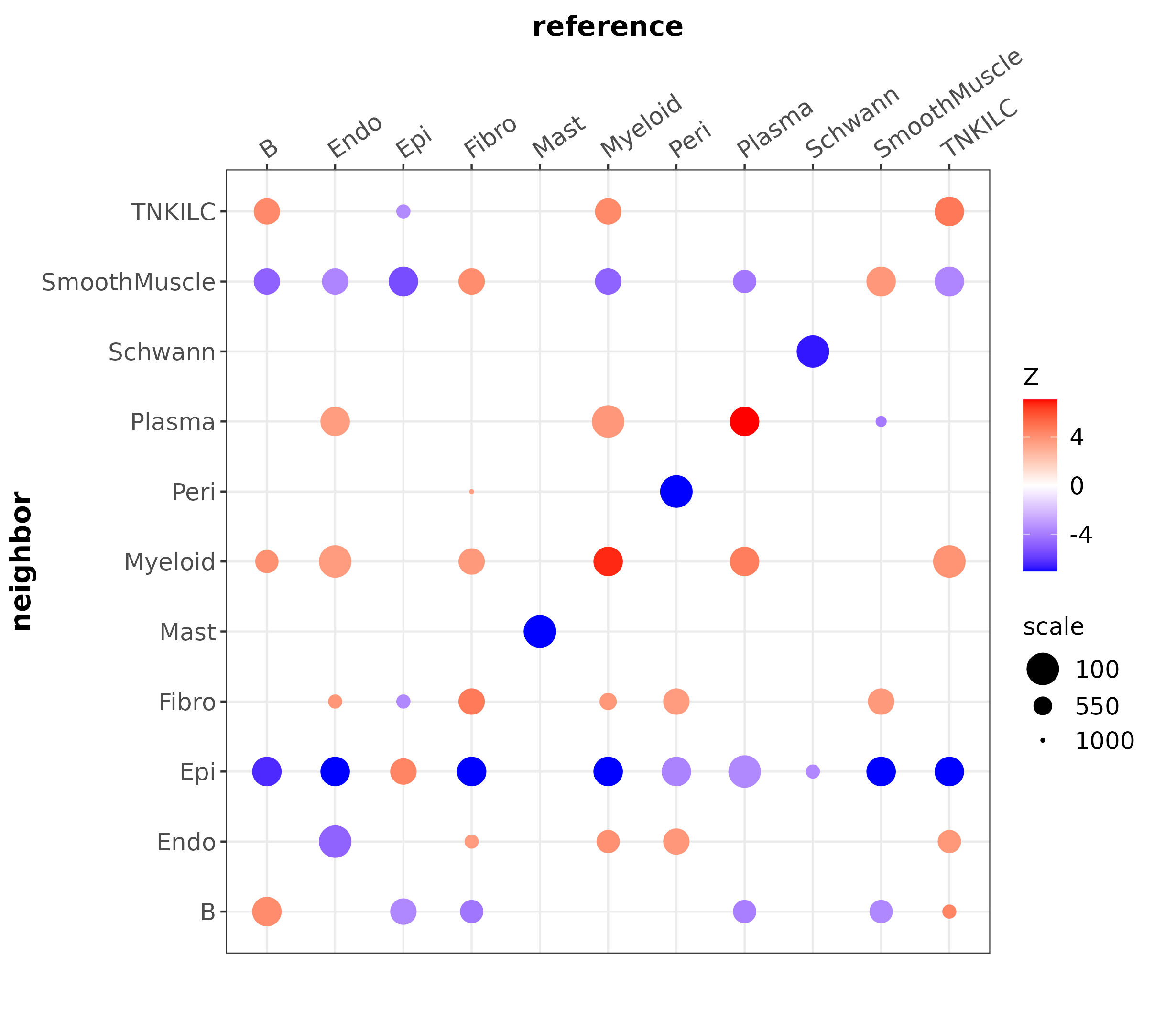
- 汇总共定位点图:
<input_name>_crawdad_summary_coloc_dotplot.png展示显著的细胞类型对共定位或互斥关系,点的大小/颜色代表强度/方向
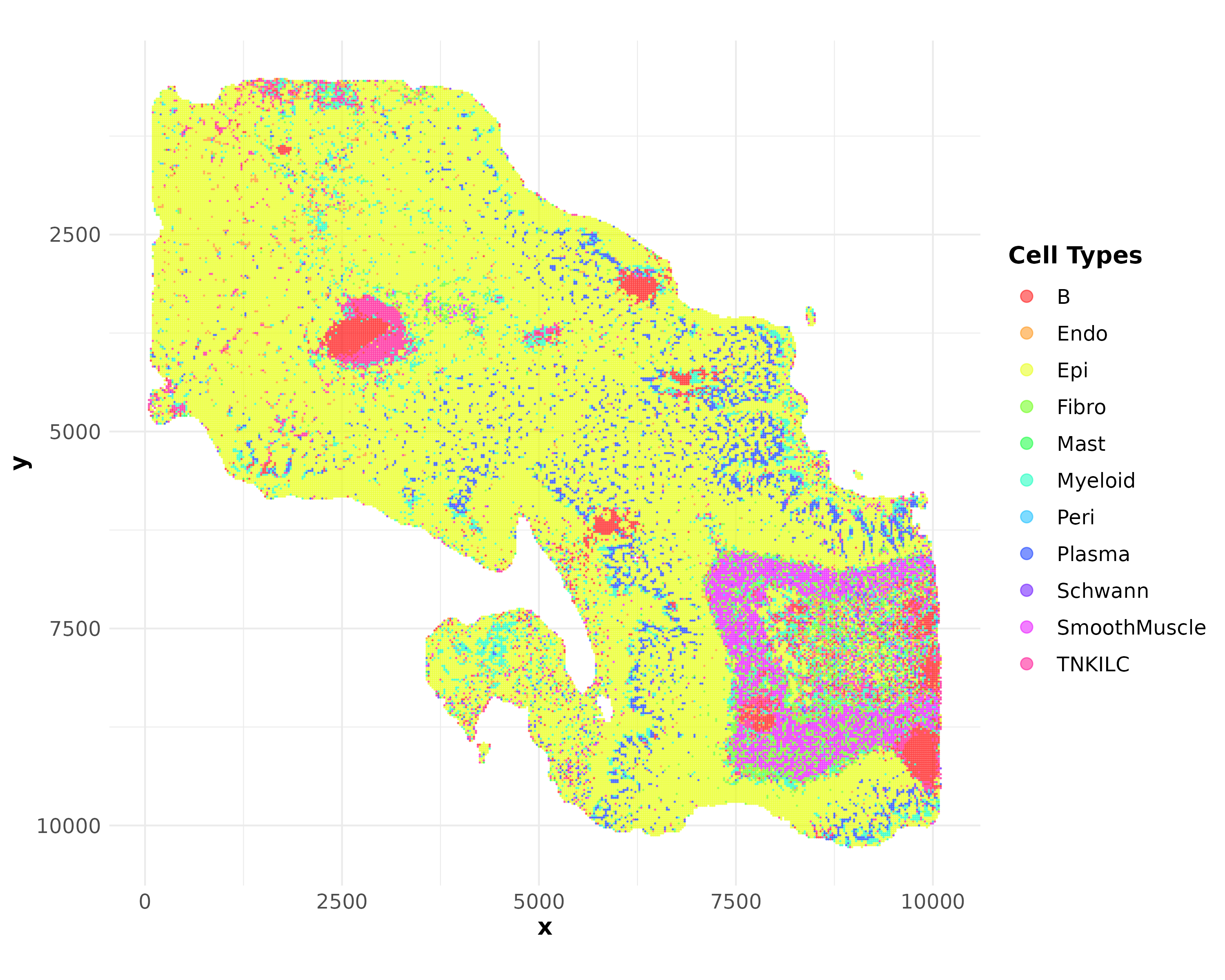
- 空间聚类图:
<input_name>_crawdad_spatial_cluster_plot.png按细胞类型着色显示各类细胞的空间排布,点大小根据数据量动态调整
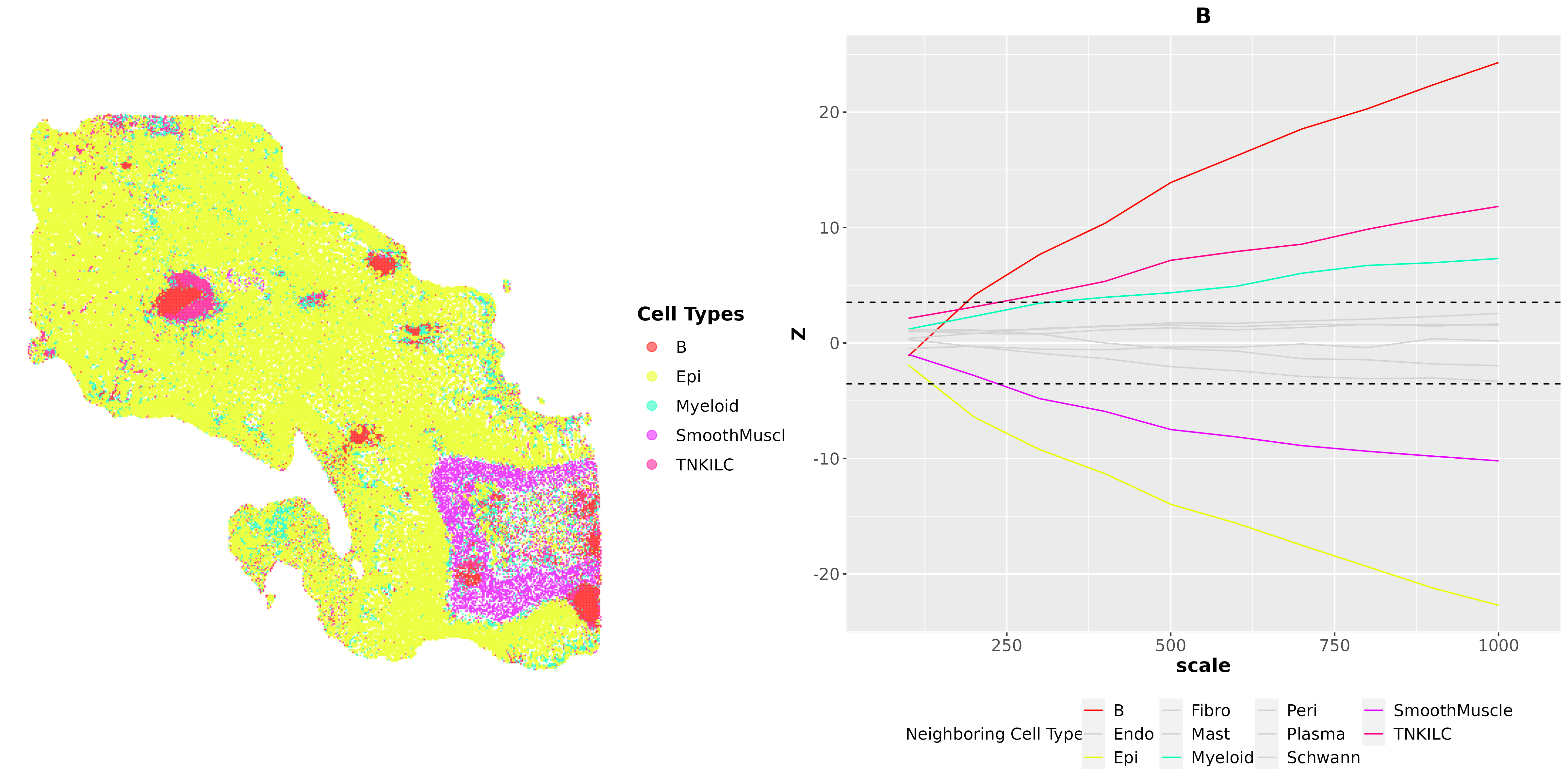
- 互作趋势图:
<input_name>_crawdad_trend_plot_ref_B.png展示B细胞与其他细胞类型的空间分布和互作强度随距离变化趋势。在趋势图中,与参考细胞类型基于 zsig 判断为显著互作的邻居细胞类型会赋予彩色,其余细胞类型则显示为浅灰色。在空间分布图中,不显著互作的细胞类型则没有显示。
结果解读说明
- Z-score:正值表示共定位(细胞类型倾向于在相同区域出现),负值表示互斥(细胞类型倾向于在不同区域出现),绝对值越大表示关系越显著
- 距离尺度:不同scale值代表不同的空间分析尺度,有助于理解细胞类型在不同空间分辨率下的相互作用模式
- 显著性阈值:zsig值用于判断统计显著性,超过此阈值的Z-score被认为是显著的空间关系
调参建议
- 若样本细胞类型较少或数据稀疏,建议调整
crawdad_perms参数以获得更稳定的结果,或调整crawdad_scales_definition以适应数据特征 crawdad_neighborhood_dist:通常设置为细胞直径的1-3倍。例如,细胞直径为20μm时,可设置为20-60μmcrawdad_perms:置换次数,增加此值会提高p值的准确性但增加计算时间,建议根据数据规模调整crawdad_scales_definition:距离尺度定义,建议根据细胞类型的大小和空间分布特征调整,起始距离应大于细胞直径spatial_coords_scale:空间坐标缩放因子,对于stereo-seq数据建议设为0.5,将坐标转为微米单位