Enrichr算法
用途与运行方式
场景一:对SDAS DEG分析得到的显著差异基因富集分析
SDAS geneSetEnrichment enrichr \ -i de_t-test.anno_rctd.SmoothMuscle-vs-Endo.sig_filtered.csv -o outdir \ --species human场景二:只用感兴趣的数据库进行显著差异基因的富集分析
SDAS geneSetEnrichment enrichr \ -i de_t-test.anno_rctd.SmoothMuscle-vs-Endo.sig_filtered.csv -o outdir \ --gmt sdas_deg_enrichment/lib/GSEADB/KEGG_2021_Human.gmt
输入参数说明
| enrichr参数 | 是否必须 | 默认值 | 描述 |
|---|---|---|---|
| -i / --input | 是 | SDAS DEG 分析得到的sig_filtered.csv文件 | |
| -o / --output | 是 | 结果存放路径 | |
| --species | 否 | human | 指定物种的数据库,'human' 或 'mouse',默认 'human',当指定--gmt参数时,该参数不起作用 |
| --gmt | 否 | gmt格式的数据库文件,其中gene name信息必须转为大写,多个文件时用','隔开,不提供时使用--species参数指定的物种数据库 | |
| --cut_off | 否 | 1 | 富集结果作图时过滤的pvalue阈值,默认值为1,设太小可能会由于没有显著富集结果导致无法作图 |
| --background | 否 | 设定富集分析时使用的background,默认为所用数据库的gene数 | |
| --top_term | 否 | 10 | 筛选top数量的通路进行作图,默认10 |
| -v / --verbose | 否 | 启用详细模式,打印任务进度。默认:False |
输出结果展示
| enrichr结果文件 | 描述 |
|---|---|
enrichment_{database}.UP.csv |
上调基因的富集分析结果 |
enrichment_{database}.DOWN.csv |
下调基因的富集分析结果 |
enrichment_{database}.pdf/png |
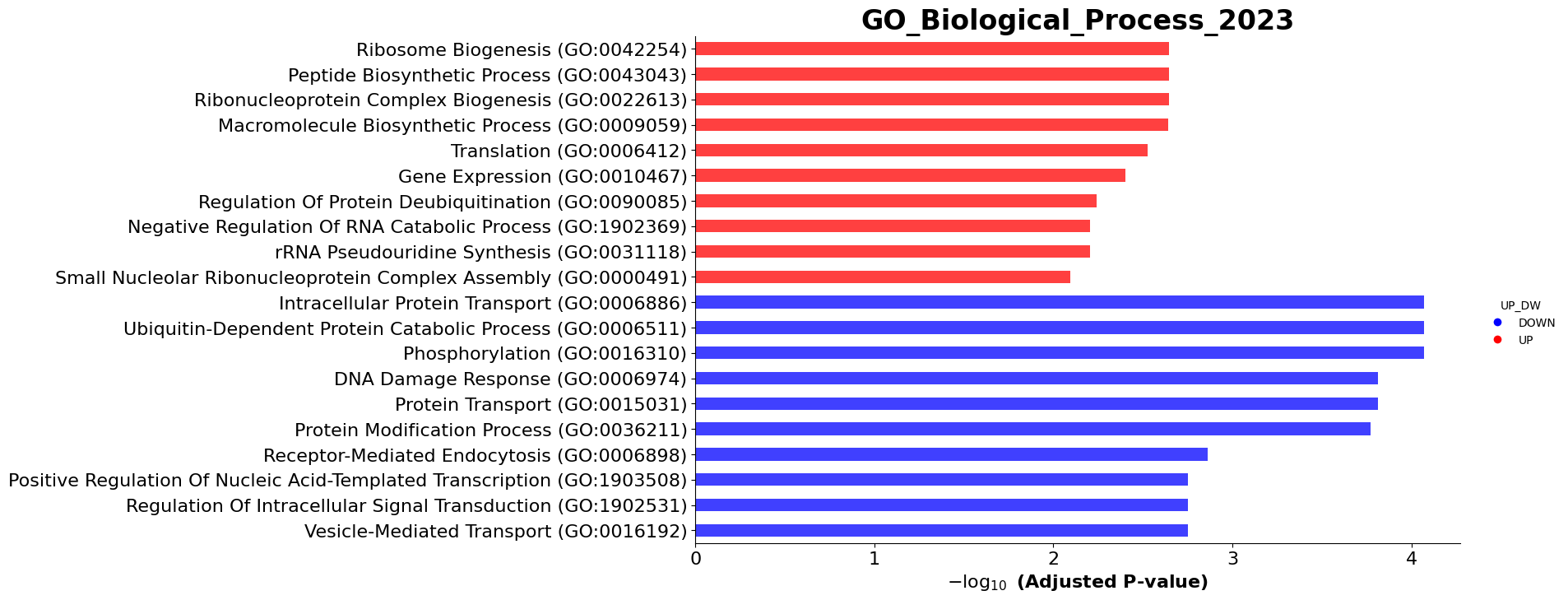
上调和下调基因显著富集通路图 |
- 上/下调基因的富集分析结果:
enrichment_{database}.UP/DOWN.csv,分别对上调和下调基因进行富集分析的结果文件,文件包含Gene_set,Term,Overlap,P-value,Adjusted P-value,Odds Ratio,Combined Score,Genes这几列,分别表示基因集所属的数据库名称、具体的功能通路名称、输入基因列表中与该基因集重叠的基因数量及比例、富集分析的原始显著性p值、校正后的p值、衡量输入基因在基因集中富集的强度值、综合评分、输入基因中与该基因集重叠的具体基因名称。
| Gene_set | Term | Overlap | P-value | Adjusted P-value | Odds Ratio | Combined Score | Genes |
|---|---|---|---|---|---|---|---|
| KEGG_2021_Human.gmt | ABC transporters | 43/45 | 0.00026880161509715636 | 0.002529897553855589 | 5.888896293211162 | 48.41577841510133 | ABCA3;ABCB4;... |
| KEGG_2021_Human.gmt | AGE-RAGE signaling pathway in diabetic complications | 90/100 | 0.00011055669162020043 | 0.0013606977430178514 | 2.928493469422023 | 26.678523151510976 | AKT1;PLCB1;... |
| KEGG_2021_Human.gmt | AMPK signaling pathway | 107/120 | 6.61066069653723e-05 | 0.0009525689822440243 | 2.709298083129859 | 26.074940028325518 | AKT1;CREB3;... |
| ... | ... | ... | ... | ... | ... | ... | ... |
- 富集分析结果条形图:
enrichment_{database}.pdf/png不同颜色分别表示上下调基因最富集的top通路。