GSEA算法
用途与运行方式
场景一:指定类别1与类别2进行gsea分析,其中
--ident1做为处理,--ident2做为对照SDAS geneSetEnrichment gsea -i st.h5ad -o outdir \ --group_key leiden --ident1 1 --ident2 2 --species human场景二:对obs的某列做子集后再进行gsea分析
SDAS geneSetEnrichment gsea -i st.h5ad -o outdir \ --group_key leiden --ident1 1 --ident2 2 --species human \ --subset_key cell_type --subset_values B场景三:只用感兴趣的数据库进行分析
SDAS geneSetEnrichment gsea -i st.h5ad -o outdir \ --group_key leiden --ident1 1 --ident2 2 \ --gmt sdas_deg_enrichment/lib/GSEADB/h.all.v2024.1.Hs.symbols.gmt,sdas_deg_enrichment/lib/GSEADB/KEGG_2021_Human.gmt场景四:只对感兴趣的通路进行作图,将感兴趣的通路全名写入一个txt文档里面,每个通路一行,然后将这个txt文档通过
--pathways参数传入分析流程。需要注意的是使用的数据库中必须包含这些指定的通路名称SDAS geneSetEnrichment gsea -i st.h5ad -o outdir \ --group_key leiden --ident1 1 --ident2 2 \ --pathways ./pathway.txt \ --gmt sdas_deg_enrichment/lib/GSEADB/h.all.v2024.1.Hs.symbols.gmt,sdas_deg_enrichment/lib/GSEADB/KEGG_2021_Human.gmt
输入参数说明
| gsea参数 | 是否必须 | 默认值 | 描述 |
|---|---|---|---|
| -i / --input | 是 | Stereo-seq h5ad,要求原始矩阵 | |
| --group_key | 是 | 需要用于进行gsva分析的对象,用','分隔多个对象 | |
| --ident1 | 是 | 用于进行GSEA分析的对象名,类似处理组 | |
| --ident2 | 是 | 用于做对照的对象名 | |
| -o / --output | 是 | 结果存放路径 | |
| --layer | 否 | 指定表达矩阵,默认用adata.raw.X或adata.X | |
| --gene_symbol_key | 否 | real_gene_name | 指定gene name所在列,默认real_gene_name,如果没有则使用矩阵中的原始基因信息 |
| --species | 否 | human | 指定数据库,'human' 或 'mouse',默认 'human',当指定--gmt参数时,该参数不起作用 |
| --subset_key | 否 | 需要提取的值所属的obs名称 | |
| --subset_values | 否 | 需要提取的信息,存在多个时用','分隔 | |
| --sample_size | 否 | 0 | 对输入文件的bin数量进行随机取样以减小内存消耗,默认值为0,不做采样 |
| --gmt | 否 | gmt格式的数据库文件,其中gene name信息必须为大写,多个文件时用','隔开 | |
| --graph | 否 | 5 | 筛选top数量的通路进行画图,默认'5',指定--pathways参数时,该参数不起作用 |
| --pathways | 否 | 通过txt文件指定一个或多个感兴趣的通路进行画图,这些通路必须在gmt数据库中 | |
| --permutation_type | 否 | gene_set | 小于1000个样本时用‘gene_set’,大于1000个样本时用‘phenotype’。默认‘gene_set’。 |
| -v / --verbose | 否 | 启用详细模式,打印任务进度。默认:False | |
| --permutation_num | 否 | 1000 | 随机置换次数(用于计算ES空值分布)。默认:1000 |
| --min_size | 否 | 15 | 基因集中包含的输入基因最小数量。默认:15 |
| --max_size | 否 | 500 | 基因集中包含的输入基因最大数量。默认:500 |
| --weight | 否 | 1 | 排序指标权重(用于调整基因权重),可选值:{0, 1, 1.5, 2}。默认:1 |
| --method | 否 | signal_to_noise | 基因排序相关性计算方法,可选值: {'signal_to_noise', 'abs_signal_to_noise', 't_test', 'ratio_of_classes', 'diff_of_classes', 'log2_ratio_of_classes'}。默认:'signal_to_noise' |
| --ascending | 否 | 设置排序指标为升序(指定此参数则ascending=True)。默认:False(降序) | |
| --seed | 否 | 123 | 随机数种子值。默认:123 |
| --threads | 否 | 1 | 并行计算使用的线程数。默认:1 |
输出结果展示
| gsea结果文件 | 描述 |
|---|---|
GSEA.{database}.csv |
csv格式的结果文件 |
GSEA.{database}.top5.pdf/png |
pdf和png格式的图像文件 |
- 文件格式示例:
GSEA.{database}.csv这个文件是gsea分析结果文件,包含了Name,Term,ES,NES,NOM p-val,FDR q-val,FWER p-val,Tag %,Gene %,Lead_genes等信息。其中Term是通路名称;ES是富集得分(Enrichment Score),反映基因集成员在排序基因列表(如差异表达基因排序)中的富集程度,正ES:基因集在排序列表顶部富集(与表型正相关),负ES:基因集在排序列表底部富集(与表型负相关);NES是标准化富集得分 (Normalized Enrichment Score);NOM p-val是名义p值;FDR q-val是校正后的p值;FWER p-val是族系错误率校正后的p值;Tag %是基因集中位于排序列表核心富集区域的基因百分比;Gene %是分析中实际使用到的基因占基因集总基因数的百分比;Lead_genes是对富集得分(ES)贡献最大的核心基因。
| Name | Term | ES | NES | NOM p-val | FDR q-val | FWER p-val | Tag % | Gene % | Lead_genes |
|---|---|---|---|---|---|---|---|---|---|
| gsea | HALLMARK_MYC_TARGETS_V1 | 0.7472938191195556 | 2.39333105644001 | 0.0 | 0.0 | 0.0 | 160/195 | 18.89% | RPL14;HNRNPA2B1;... |
| gsea | HALLMARK_OXIDATIVE_PHOSPHORYLATION | 0.7431758291176868 | 2.376055485647371 | 0.0 | 0.0 | 0.0 | 168/200 | 20.44% | MDH2;COX8A;... |
| gsea | HALLMARK_ALLOGRAFT_REJECTION | 0.744882727767552 | 2.3688992213810462 | 0.0 | 0.0 | 0.0 | 118/194 | 14.03% | ITGB2;HLA-DRA;... |
| gsea | ... | ... | ... | ... | ... | ... | ... | ... | ... |
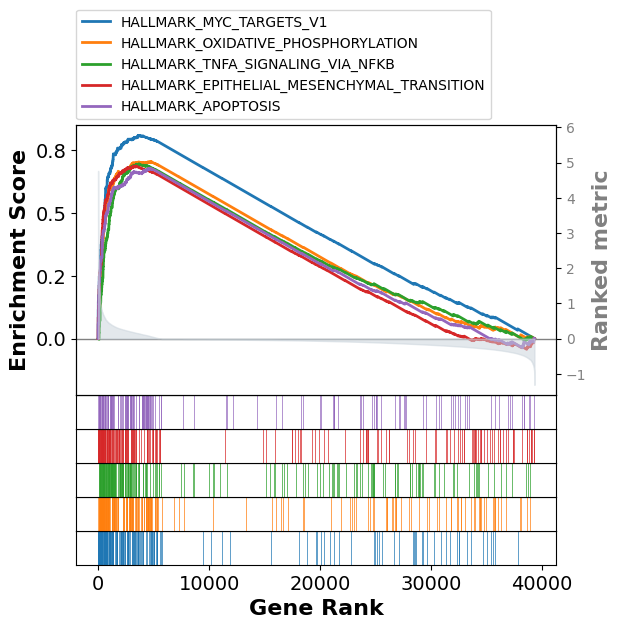
- top Terms富集曲线图:
GSEA.{database}.top5.pdf/png(见下图示例),图中Enrichment Score为正说明改Term与--ident1正相关,为负说明为负相关。