graphST算法
运行方式
SDAS spatialDomain graphst –i st.h5ad –o outdir --n_clusters 10 --bin_size 100 \
--gpu_id 1 --n_hvg 3000
输入参数说明
| 参数 | 是否必须 | 默认值 | 描述 |
|---|---|---|---|
| -i / --input | 是 | Stereo-seq h5ad,要求有原始表达矩阵 | |
| -o / --output | 是 | 输出文件夹 | |
| --n_clusters | 是 | 聚类数目 | |
| --bin_size | 是 | Bin大小,用于控制图中点的大小,不用于计算,比如20,50,100, cellbin (等效于20) | |
| --tool | 否 | mclust | Graphst所选的聚类方法,可选'mclust', 'leiden', louvain' |
| --layer | 否 | 指定Stereo-seq h5ad存放raw counts的layer | |
| --n_hvg | 否 | 3000 | 使用的高变基因数 |
| --slice_key | 否 | 多片h5ad.obs中表示片编号的列的名称,如果是多片,必须指定* | |
| --gpu_id | 否 | -1 | 使用的GPU的编号,如果为-1,则使用CPU |
| --seed | 否 | 42 | 随机种子设置 |
*符合下列情况,可以进行多片分析:
- 邻片,组织纵向切片,做空间域分析前,需要进行邻片配准,参数slice_key设置为h5ad的obs中记录多片的key。
- 同一个组织切片,在水平方向上分成若干连续多片,分开测序,需要多片在水平方向上配准。
具体可参考:
- https://deepst-tutorials.readthedocs.io/en/latest/Tutorial%204\_Horizontal%20Integration.html (邻片)
- https://deepst-tutorials.readthedocs.io/en/latest/Tutorial%205\_Vertical%20Integration.html (水平方向)
输出结果展示
| 结果文件 | 描述 |
|---|---|
<input_name>_domain_graphst.h5ad |
输入h5ad+空间域聚类结果,聚类结果保存在obs['domain_graphst']中 |
<input_name>_domain_graphst.png/pdf |
空间域cluster在组织上的分布图,同时输出png和pdf |
<input_name>_domain_graphst_split.png/pdf |
空间域不同cluster分开展示的图,同时输出png和pdf |
- 空间域cluster在组织上的分布图:
<input_name>_domain_graphst.png/pdf颜色代表每个bin/cellbin的空间结构域类型
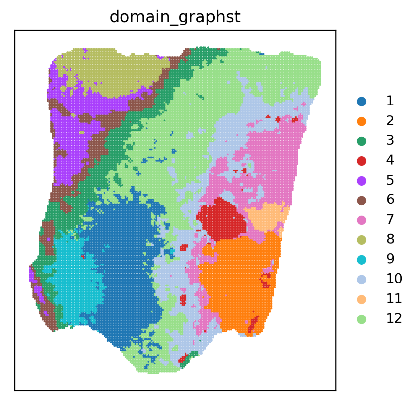
- 空间域cluster在组织上分开的分布图:
<input_name>_domain_graphst_split.png/pdf颜色代表每个bin/cellbin的空间结构域类型,标题为空间结构域类型(细胞个数)