hdWGCNA算法
用途
使用hdWGCNA 算法进行空间基因共表达基因集识别
运行方式
SDAS coexpress hdwgcna -i st.h5ad -o outdir --bin_size 100 \
--input_layer raw_counts \
--selected_genes top5000 \
--moran_path ./moran.csv \
--n_cpus 8 \
--seed 42 \
--knn_neighbors 50 \
--max_shared_cells 15 \
--soft_power 8
输入参数说明
| 参数 | 是否必须 | 默认值 | 描述 |
|---|---|---|---|
| -i / --input | 是 | Stereo-seq h5ad,要求有原始表达矩阵 | |
| -o / --output | 是 | 输出文件夹 | |
| --bin_size | 是 | 50 | 分辨率Bin大小(20, 50, 100, 200, cellbin),与输入h5ad一致 |
| --layer | 否 | 指定h5ad中原始表达矩阵的layer层 (例如layers[‘raw_counts’] | |
| --selected_genes | 否 | top5000 | 基因列表(topn高变基因, full全部基因) |
| --moran_path | 否 | 已计算好的基因莫兰指数列表路径 | |
| --n_cpus | 否 | 8 | 并行计算进程数 |
| --seed | 否 | 42 | 随机种子 |
| --knn_neighbors | 否 | 50 | 合并细胞时KNN算法覆盖的邻域spot/细胞个数(k neighbor) |
| --max_shared_cells | 否 | 15 | 合并细胞时KNN算法覆盖的最多交集spot/细胞个数 |
| --soft_power | 否 | 网络构建时使用,默认自动选择无标度拓扑模型拟合度达到 0.8 的最低 soft_power |
输出结果展示
| 结果文件 | 描述 |
|---|---|
<input_name>_hdwgcna.module.csv |
空间高变基因(gene symbol+gene id)对应的共表达基因集(module)的共表达基因集的结果csv |
<input_name>_hdwgcna.module_score.csv |
共表达基因集的基因集打分结果csv |
<input_name>_hdwgcna.coexpress.rds |
含有共表达基因集结果的rds文件 |
<input_name>_hdwgcna.module_score.png/pdf |
共表达基因集的基因集打分空间热图 |
<input_name>_hdwgcna.all_coex_dendrogram.png/pdf |
共表达基因集的相似性树状图 |
<input_name>_hdwgcna.softpowers.png/pdf |
构建网络的soft_power阈值图 |
<input_name>_hdwgcna.moran.csv |
如果使用topn计算,输出全部基因的莫兰指数以及P值 |
- 共表达基因集的结果csv:
<input_name>_hdwgcna.module.csv,以逗号分隔。hdWGCNA输出结果展示识别的空间高变基因对应的共表达基因集(module),kME表示某个基因的表达模式与所在模块的模块特征基因(Module Eigengene, ME)之间的相关性强度,kME值越接近1或-1,表明该基因越可能是枢纽基因(hub gene)
| real_gene_name | geneid | Module | color | kME_Module1 | kME_Module2 | kME_grey | kME_Module3 | kME_Module4 | kME_Module5 | kME_Module6 | kME_Module7 | kME_Module8 | kME_Module9 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A2M | ENSG00000175899 | Module1 | green | 0.47946868988301 | -0.107096403482606 | -0.178114022165641 | 0.0676792398874597 | 0.095966109797419 | -0.0907050325056857 | -0.0529390531160642 | -0.150612945887371 | 0.0878907827651177 | 0.0249952108382643 |
| A2M-AS1 | ENSG00000237094 | Module1 | green | 0.54370397007705 | -0.150011910577089 | -0.254597937099371 | 0.0926882061841318 | 0.140032173496191 | -0.115227951266487 | -0.101675353602963 | -0.222107282189061 | 0.0803636102659976 | 0.0426306888623326 |
| A2ML1 | ENSG00000166535 | Module2 | yellow | 0.0404144692736028 | 0.479908573141937 | 0.194701680726881 | -0.327610748128114 | 0.0430624759042059 | 0.429681007497005 | -0.342984504779987 | 0.145625804577339 | -0.386999928188458 | 0.08281144751312791 |
| A2MP1 | ENSG00000256069 | grey | grey | -0.046660656715667 | 0.20294339804614 | 0.284819067476003 | -0.0506850476403686 | -0.205976941174478 | 0.244779685854094 | 0.000250607520833238 | 0.170101997387916 | -0.0177549796818324 | 0.0639042087827032 |
- 共表达基因集的基因集打分结果csv:
<input_name>_hdwgcna.module_score.csv,以逗号分隔。hdWGCNA输出结果展示识别的每个共表达基因集(module)的表达量打分的高低
| Module6 | Module3 | Module8 | Module2 | grey | Module7 | Module5 | Module9 | Module1 | Module4 | |
|---|---|---|---|---|---|---|---|---|---|---|
| 2200_16100 | -3.23688863476392 | -4.34756288337066 | -2.3278151796256 | -8.21694142422341 | -14.8112682710791 | -9.12253218247156 | -10.174563894144 | -3.09447240000024 | 0.481660736850741 | 3.91787079378259 |
| 2200_17200 | 5.77873502485046 | 0.783016254503074 | 1.06582091429724 | -6.03050203635639 | -3.71256039305597 | -0.825856084852031 | -3.67468239887104 | -2.09159016878048 | -2.639251117267012 | 5.41583186417414 |
| 2300_16700 | 7.90521666109811 | 2.93759207152763 | -0.391450035802177 | -3.02639637030598 | 1.63013439679168 | 1.66371621513915 | -1.51360146647437 | -0.8975499248414 | -4.66703690157902 | 1.40723191567521 |
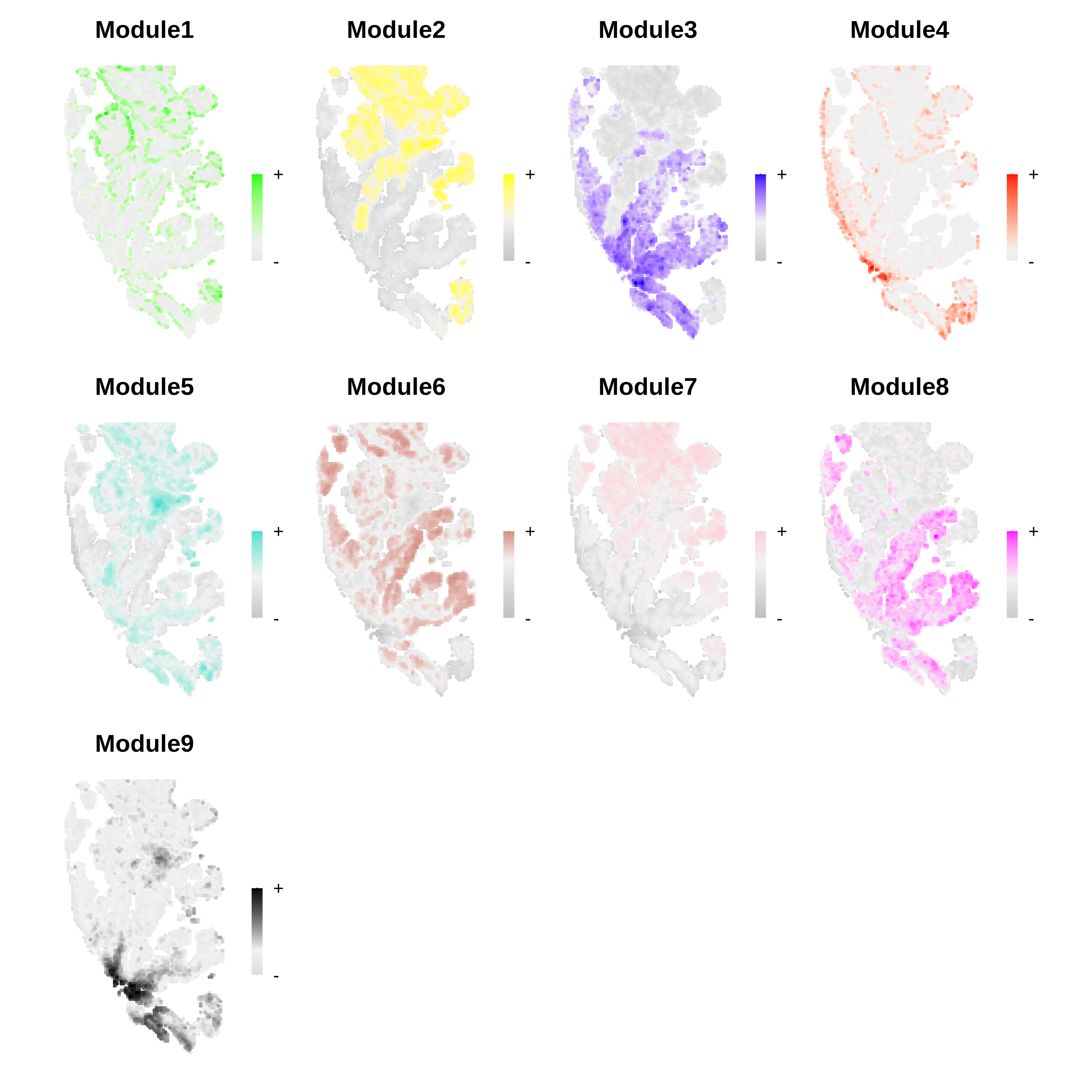
- 共表达基因集的基因集打分空间热图
<input_name>_hdwgcna.module_score.png/pdf:可视化所有共表达基因集(Module)的空间分布模式。图中颜色强度表示共表达基因集表达量的高低
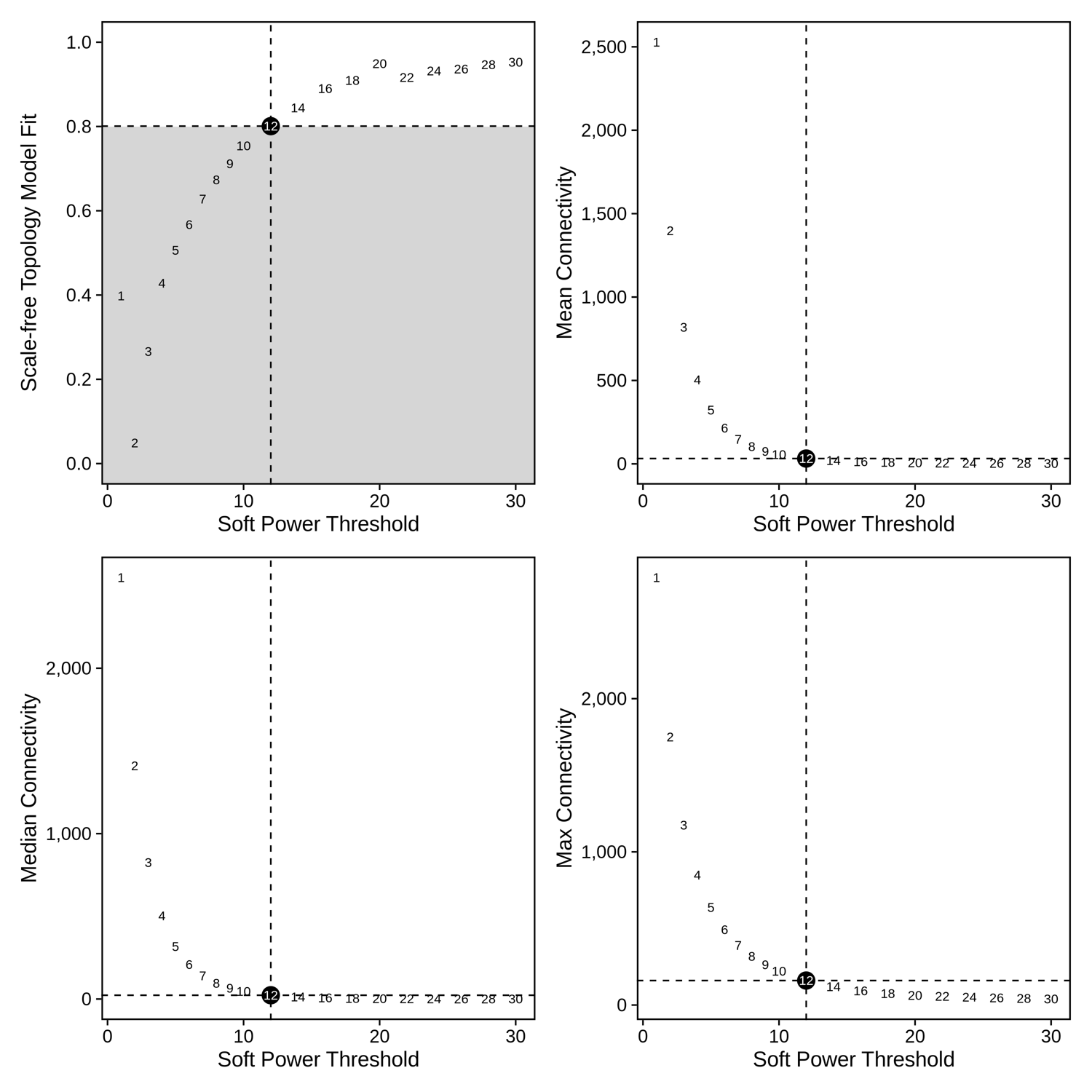
- 构建网络的soft_power阈值图
<input_name>_hdwgcna.softpowers.png/pdf:分析不同软阈值参数的网络构建效果,默认自动选择无标度拓扑模型拟合度达到 0.8 的最低 soft_power
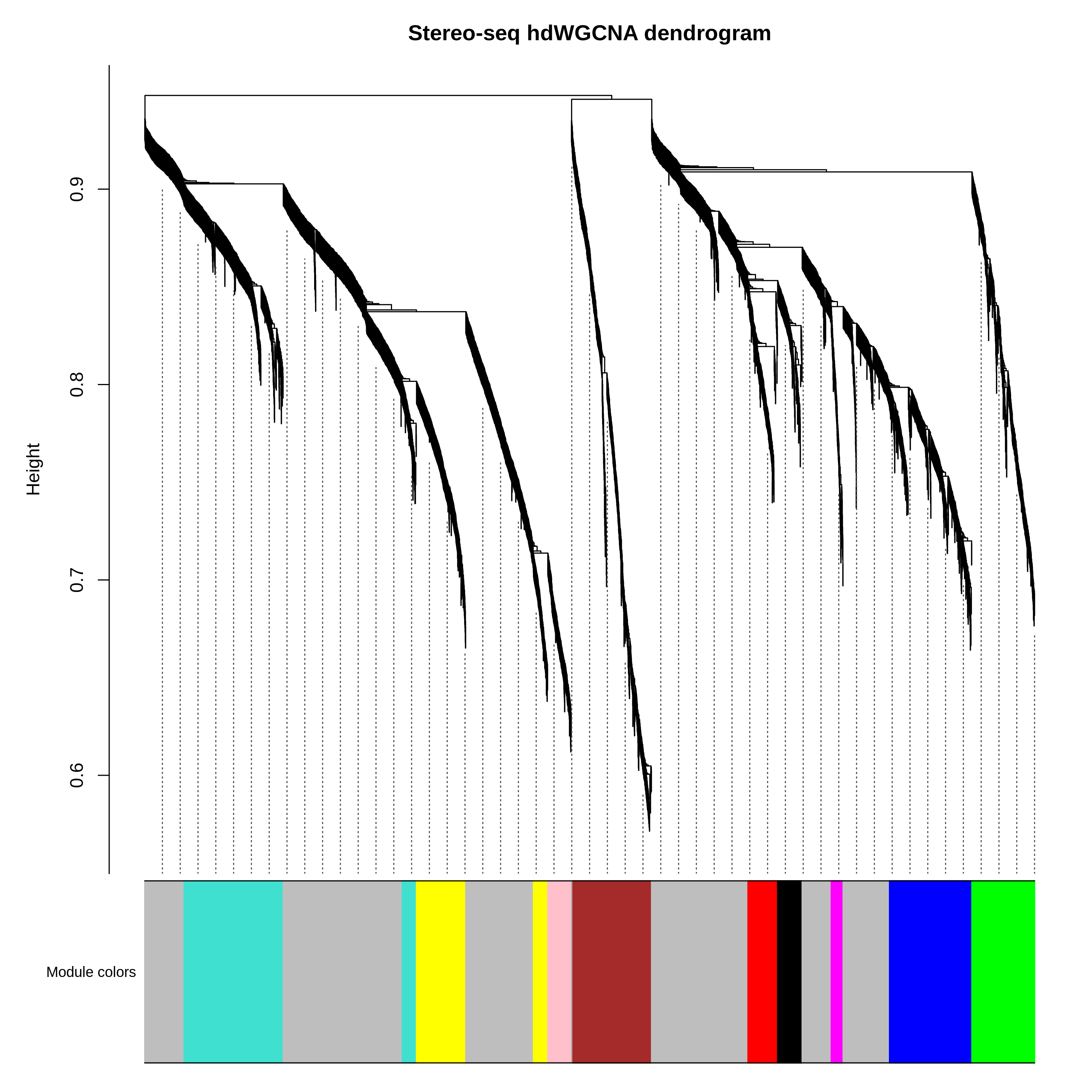
- 共表达基因集的相似性树状图
<input_name>_hdwgcna.all_coex_dendrogram.png/pdf:展示不同共表达基因集(Module)之间的相似性聚类树状图
结果解读说明
- 共表达基因集从Module1开始,grey为不符合共表达基因集聚类要求的基因。
调参建议
- 若样本bin20/50基因数低于200,或其他特殊样本,识别的空间共表达基因集较少,可根据
soft_power测试图调低阈值。 - 可自定义
knn_neighbors、max_shared_cells参数以获得更多可解读结果。