NeST算法
用途
使用NeST 算法进行空间基因共表达基因集识别
运行方式
SDAS coexpress nest -i st.h5ad -o outdir --bin_size 100 \
--layer raw_counts \
--selected_genes top5000 \
--moran_path ./moran.csv \
--n_cpus 8 \
--seed 42 \
--hotspot_min_size 30 \
--hotspot_min_samples 4 \
--min_cells 100
输入参数说明
| 参数 | 是否必须 | 默认值 | 描述 |
|---|---|---|---|
| -i / --input | 是 | Stereo-seq h5ad,要求有原始表达矩阵 | |
| -o / --output | 是 | 输出文件夹 | |
| --bin_size | 是 | 50 | 分辨率Bin大小(20, 50, 100, 200, cellbin),与输入h5ad一致,画图与计算均需要 |
| --layer | 否 | 指定h5ad中原始表达矩阵的layer层 (例如layers[‘raw_counts’] | |
| --selected_genes | 否 | top5000 | 基因列表(topn高变基因, full全部基因) |
| --moran_path | 否 | 已计算好的基因莫兰指数列表路径 | |
| --n_cpus | 否 | 8 | 并行计算进程数 |
| --seed | 否 | 42 | 随机种子 |
| --hotspot_min_size | 否 | 30 | 空间高变单基因覆盖的最少spot/细胞数 |
| --hotspot_min_samples | 否 | 4 | 识别空间高变单基因时DBSCAN算法覆盖的最少邻域spot/细胞个数(k neighbor) |
| --min_cells | 否 | 100/30 | 共表达基因集覆盖的最少spot/细胞个数,默认:cellbin/bin20/bin50时为100;bin100/bin200时为30 |
输出结果展示
| 结果文件 | 描述 |
|---|---|
<input_name>_nest.module.csv |
空间高变基因(gene symbol+gene id)对应的共表达基因集(module)的共表达基因集的结果csv |
<input_name>_nest.h5ad |
含有共表达基因集结果的h5ad文件(adata.obsm[‘module_score_nest’]) |
<input_name>_nest_module_score_nest.png/pdf |
共表达基因集的基因集打分空间热图 |
<input_name>_nest.all_coex_hotspots/_nest.all_coex_structure.png/pdf |
共表达基因集的空间位置与层级结构 |
<input_name>_nest.separate_coex_hotspots.png/pdf |
共表达基因集的空间位置与基因个数 |
<input_name>_nest.moran.csv |
如果使用topn计算,输出全部基因的莫兰指数以及P值 |
- 共表达基因集的结果csv:
<input_name>_nest.module.csv,以逗号分隔。NeST识别的空间高变基因对应的共表达基因集(module)
| Module | geneid | real_gene_name |
|---|---|---|
| Module0 | ENSG00000116016 | EPAS1 |
| Module0 | ENSG00000106554 | CHCHD3 |
| Module0 | ENSG00000104164 | MDGA2 |
- 共表达基因集的基因集打分空间热图
<input_name>_nest_module_score_nest.png:可视化所有共表达基因集(Module)的空间分布模式。图中颜色强度表示共表达基因集表达量的高低
.png)
- 共表达基因集的空间位置与层级结构
<input_name>_nest.all_coex_hotspots.png/pdf;<input_name>_nest.all_coex_structure.png/pdf:展示不同共表达基因集(Module)之间的层级关系。图中颜色表示不同共表达基因集所在的空间区域
.png)
- 共表达基因集的空间位置与基因个数
<input_name>_nest.separate_coex_hotspots.png/pdf:可视化所有共表达基因集(Module)所在的空间区域以及包含的基因数量
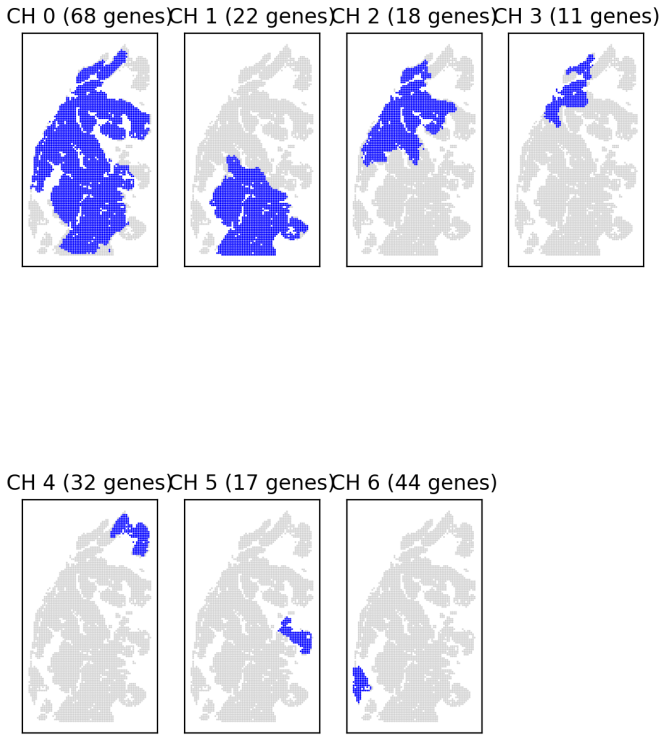
结果解读说明
- 共表达基因集从Module0开始,没有Module为不符合共表达基因集聚类要求的基因。
调参建议
- 若样本bin20/50基因数低于200,或其他特殊样本,识别的空间高变基因较少,建议降低
hotspot_min_size到10。 - 识别的空间共表达基因集较少,建议降低
min_cells到10。 - 若识别pattern过于精细,出现"NumPy Unable to allocate X GiB array"报错,建议升高
hotspot_min_size和hotspot_min_samples。