InferCNV算法
用途
利用inferCNV工具对空间转录组数据进行拷贝数变异(CNV)推断,帮助揭示组织中不同区域的基因组变异特征。同时该工具也支持对scRNA-Seq数据进行CNV推断。
运行方式
- 使用细胞注释后的h5ad,准备rds文件:
SDAS dataProcess h5ad2rds -i st.h5ad -o outdir
- 运行inferCNV
SDAS infercnv -i st.rds --h5ad st.h5ad --bin_size 50 --slice_key batch \
-o outdir --label_key anno_cell2location --species human \
--ref_group_names B,T --min_counts_per_cell 200
输入参数说明
| 参数 | 是否必须 | 默认值 | 描述 |
|---|---|---|---|
| --run_mode | 否 | stRNA | 选择空间转录组(stRNA)或单细胞(scRNA)模式 |
| -i / --input | 是 | rds文件,要求有原始表达矩阵 | |
| -o / --output | 是 | 所有文件的输出目录 | |
| --h5ad | 是 | h5ad格式的sample.h5ad,用于空间热图(单细胞模式无需设置) | |
| --bin_size | 是 | Bin大小,控制空间热图点大小(如20,50,100),单细胞模式无需设置 | |
| --label_key | 是 | rds对象metadata中的注释信息字段 | |
| --ref_group_names | 否 | 参考分组名,指定normal cell/sample分组,默认用全部cell(不推荐) | |
| --gene_order_file | 否 | 用户自定义基因染色体位置信息文件,不提供时,使用--species指定的软件自带文件。 | |
| --cluster_heatmap | 否 | False | 是否对cell聚类展示CNV heatmap(True/False) |
| --species | 否 | human | 指定预先构建好的物种的*_pos.txt,‘human’ 或者‘mouse’,默认‘human’,当指定--gene_order_file参数时,该参数不起作用 |
| --slice_key | 否 | sampleID | 多片h5ad.obs中表示片编号的列名 |
| --gene_symbol_key | 否 | real_gene_name | rds中基因名(symbol)的列名(_index为矩阵列名) |
| --assay | 否 | rds中用于计算cnv的assay名称 | |
| --cutoff | 否 | 0.02 | 基因过滤参数,参考细胞/bins中每基因最小平均counts数 |
| --min_counts_per_cell | 否 | 100 | 每个cell的最小counts数 |
输出结果展示
| 结果文件 | 描述 |
|---|---|
<input_name>_run.final.infercnv_obj.rds |
包含所有基因和spot的cnv矩阵的rds对象 |
<input_name>_CNV_score.csv |
每个spot的cnv score |
<input_name>_CNV_ref.png/pdf |
参考细胞的CNV表达热图(ref_group_names为None时不输出) |
<input_name>_CNV_obs.png/pdf |
观测细胞的CNV表达热图 |
<input_name>_CNV_score.png/pdf |
CNV score的空间热图(多片时每片一张,单细胞模式不输出) |
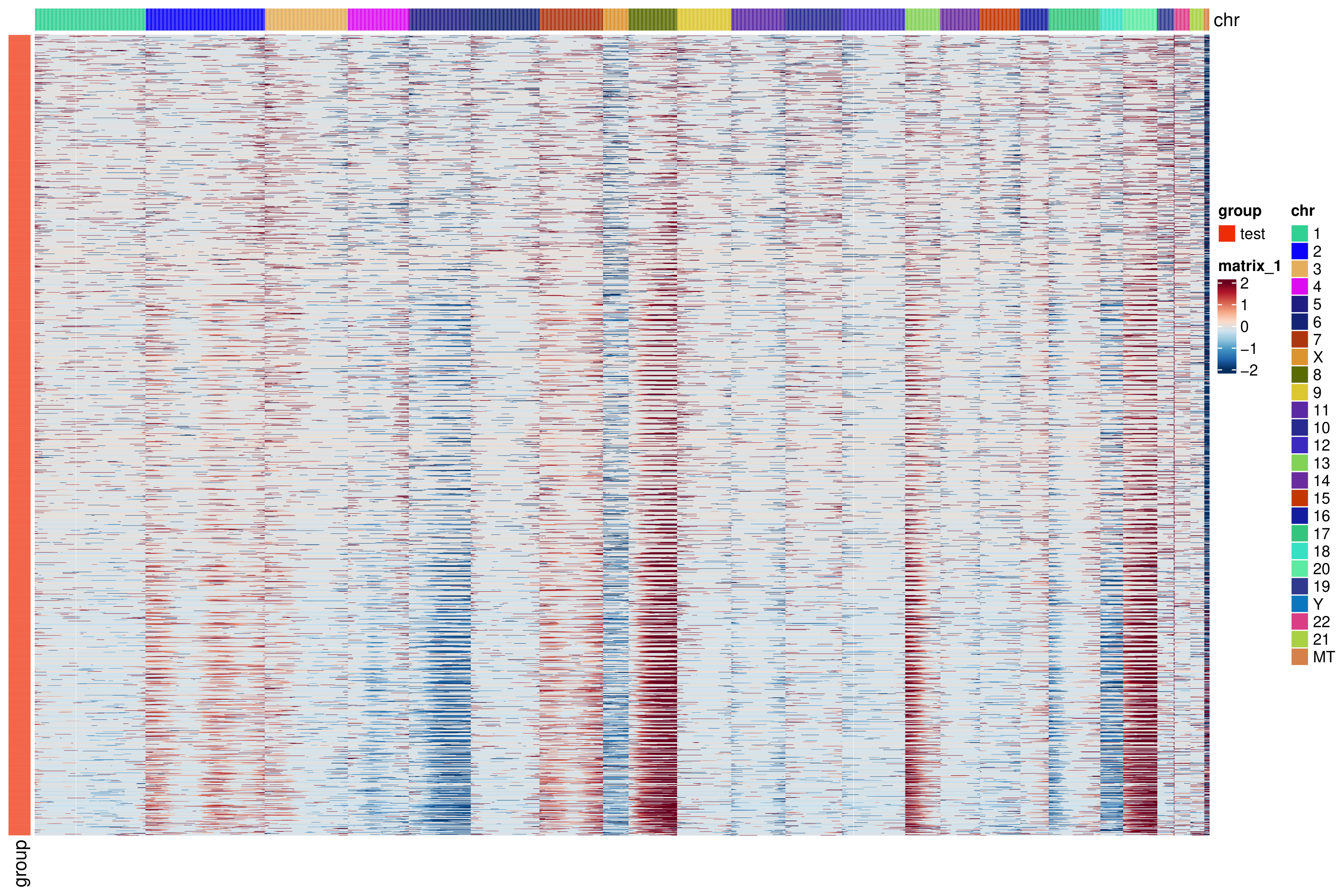
- 参考细胞的CNV表达热图:
<input_name>_CNV_ref.png/pdf横轴为spot,纵轴为基因,颜色为CNV强度

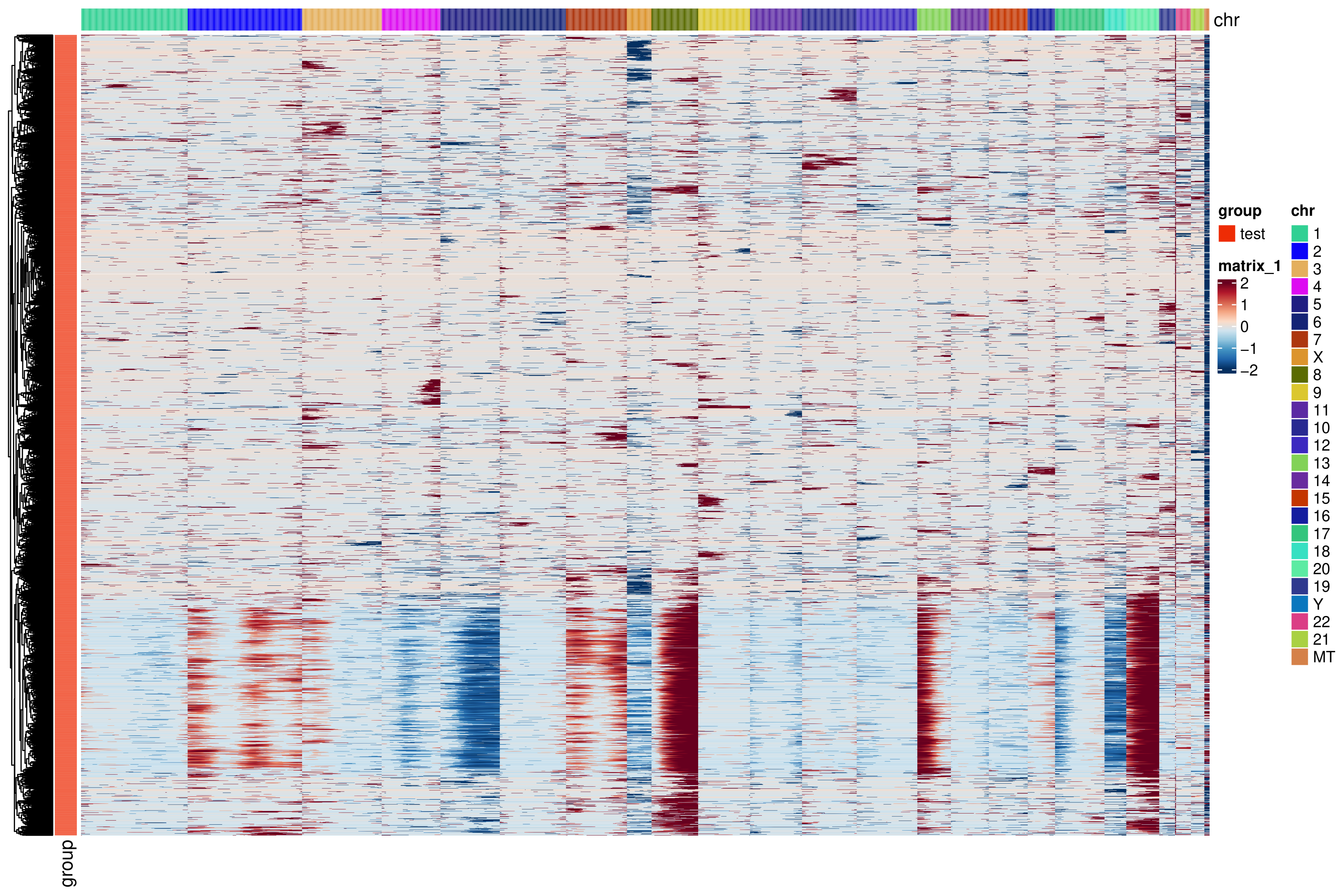
- 观测细胞的CNV表达热图:
<input_name>_CNV_obs.png/pdf横轴为spot,纵轴为基因,颜色为CNV强度
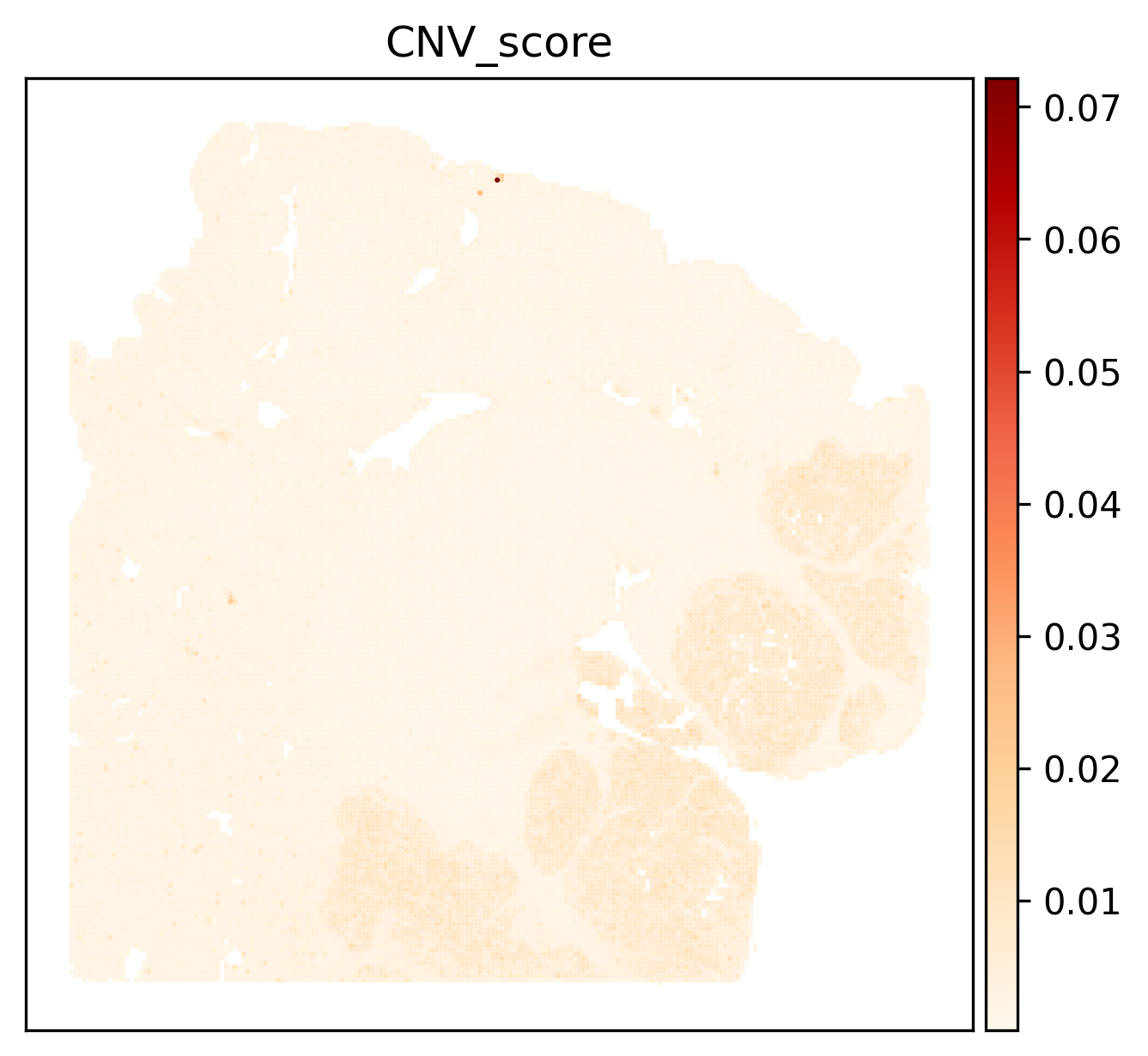
- CNV score空间热图:
<input_name>_CNV_score.png/pdf颜色为CNV强度
- CNV score文本文件:
<input_name>_CNV_score.csv,值越大代表CNV强度越高
| spot_id | CNV_score |
|---|---|
| 429496737600_D03663C6 | 0.0018 |
| 429496737700_D03663C6 | 0.0015 |
| 429496737800_D03663C6 | 0.0031 |
| ... | ... |