cellphonedb算法
用途
使用cellphonedb进行细胞通讯分析
运行方式
SDAS CCI cellphonedb -i st.h5ad --label_key anno_spotlight -o outdir \
--method statistical \
--counts_data hgnc_symbol \
--species human
输入参数说明
| 参数 | 是否必须 | 默认值 | 描述 |
|---|---|---|---|
| -i / --input | 是 | h5ad文件,要求有原始表达矩阵 | |
| -o / --output | 是 | 输出文件夹 | |
| --label_key | 是 | 细胞类群的列名 | |
| --method | 否 | statistical | 分析方法,可选simple,statistical,degs |
| --layer | 否 | h5ad存有原始表达矩阵的layer名称 | |
| --counts_data | 否 | hgnc_symbol | adata.var.index类型,可选hgnc_symbol,ensembl,gene_name |
| --threshold | 否 | 0.1 | 表达特定配体/受体的细胞百分比阈值,默认0.1即10% |
| --species | 否 | human | SDAS内置的cellphonedb的human数据库,数据库版本为v5.0.0, 当指定--cellphonedb_databa时,该参数不起作用 |
| --cellphonedb_database | 否 | 用户自定义cellphonedb数据库文件路径 | |
| --pvalue | 否 | 0.05 | 显著性阈值(筛选p≤pvalue的互作对),method为statistical时的参数 |
| --subsampling | 否 | 是否降采样,subsampling,method为statistical时的参数 | |
| --subsampling_num_cells | 否 | 降采样细胞数,默认为1/3 cells,使用--subsampling时生效,method为statistical时的参数 | |
| --microenvs_file_path | 否 | 微环境文件路径(两列:细胞类型、空间微环境) | |
| --active_tfs_file_path | 否 | 活性转录因子文件路径(两列:细胞类型、TF) | |
| --degs_file_path | 否 | 差异表达基因文件路径(两列:细胞类型、差异基因),method为degs时必须提供 | |
| --n_cpus | 否 | 8 | 线程数 |
| --seed | 否 | 42 | 随机种子 |
输出结果展示
| 结果文件 | 描述 |
|---|---|
|
所有配体-受体对的平均表达量是矩阵。每一行为一个配体-受体对,每一列为细胞类型组合,数值为平均表达量 |
|
各细胞类型的平均表达量,用于展示每个细胞类型的表达情况 |
|
各细胞类型的表达比例,反映每个配体-受体对在不同细胞类型中的表达百分比 |
|
细胞类型间交互强度评分,数值越大表示细胞间的配受体互作越强 |
|
显著交互的平均表达量 |
statistical_analysis_pvalues_<input_name>.txt |
statistical方法下,所有配体-受体对的显著性p值矩阵 |
cellphonedb_heatmap_<input_name>.png/pdf |
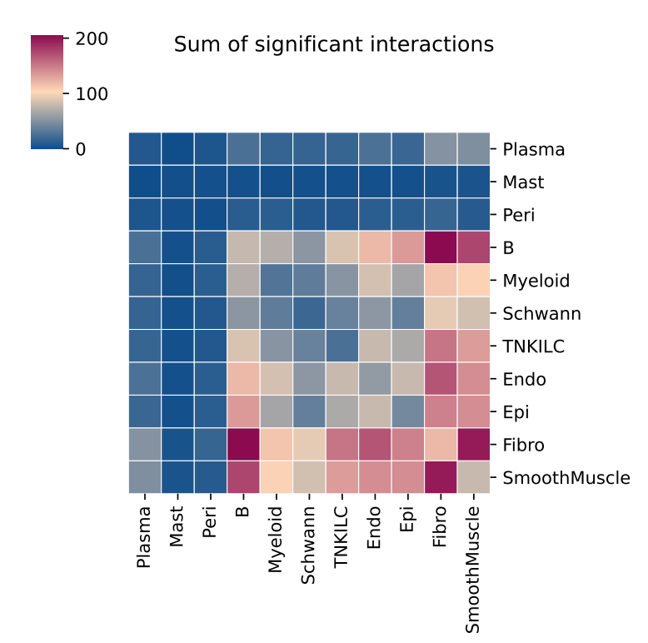
显著交互热图,展示所有细胞类群间显著配受体互作的强度,颜色深浅反映交互强度,当method为statistical和degs时输出 |
- cellphonedb输出的显著互作热图:
cellphonedb_heatmap_<input_name>.png/pdf展示所有细胞类群间显著配受体互作的热图,颜色深浅反映交互强度。 横轴和纵轴均为细胞类群,颜色深浅表示配受体互作强度,颜色越深表示互作越强。可快速比较不同细胞类群间的通讯活跃程度