构建单细胞参考数据
用途
使用cell2locationMakeRef构建cell2location的单细胞参考inf_aver.csv文件
运行方式
SDAS cellAnnotation cell2locationMakeRef -o ./ref --reference sc.h5ad --label_key annotation \
--batch_key id \
--nonz_mean_cutoff 1.45 \
--gpu_id 3
输入参数说明
| 参数 | 是否必须 | 默认值 | 描述 |
|---|---|---|---|
| -o / --output | 是 | 输出文件夹 | |
| --reference | 是 | 单细胞ref h5ad,要求有原始表达矩阵 | |
| --label_key | 是 | 单细胞ref h5ad.obs中表示细胞类型的列的名称 | |
| --ref_layer | 否 | 单细胞ref h5ad存放raw counts的layer | |
| --ref_gene_symbol_key | 否 | _index | 单细胞ref h5ad.var中表示基因名(symbol)的列的名称 (_index 表示使用h5ad.var.index) |
| --batch_key | 否 | 单细胞ref h5ad.obs中表示批次的列的名称,不输入则不考虑批次 | |
| --filter_rare_cell | 否 | 100 | 如果某些细胞类型在单细胞ref中细胞数小于此值,则过滤掉这些细胞类型 |
| --check_filter_genes | 否 | 如果设置此参数,则只输出筛选基因的结果图filter_genes.png | |
| --cell_count_cutoff | 否 | 5 | 控制cell2location筛选基因的参数,一般不调整 |
| --cell_percentage_cutoff2 | 否 | 0.03 | 控制cell2location筛选基因的参数,值越大筛选出的基因越少,基因数推荐控制在8k-16k |
| --nonz_mean_cutoff | 否 | 1.12 | 控制cell2location筛选基因的参数,值越大筛选出的基因越少,基因数推荐控制在8k-16k |
| --max_epochs | 否 | 250 | 模型训练epoch数 |
| --seed | 否 | 42 | 随机种子设置 |
| --gpu_id | 否 | -1 | 使用的GPU的编号,如果为-1,则使用CPU。 此参数只指定主要使用的GPU,其他GPU也会被占用,但占用量很低。如果需要严格指定GPU,请在运行前设置环境变量,如: export CUDA_VISIBLE_DEVICES=2,此时再设置--gpu_id 0,则会只使用2号GPU |
| --n_threads | 否 | CPU模式下使用的线程数,默认为全部CPU |
输出结果展示
| 结果文件 | 描述 |
|---|---|
<reference_name>_filter_genes.png/pdf |
Cell2location筛选基因的结果图 (\ |
<reference_name>_train_history.png/pdf |
训练Loss下降图 |
<reference_name>_inf_aver.csv |
Cell2location构建的单细胞ref csv |
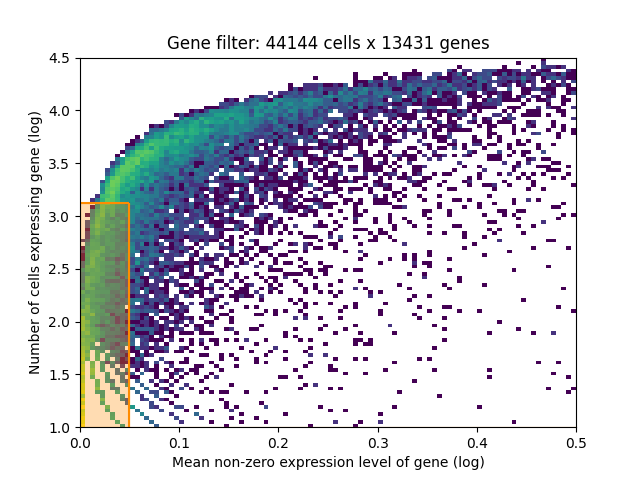
- Cell2location筛选基因的结果图:
<reference_name>_filter_genes.png/pdf橙色区域标出了根据表达该基因的细胞数 (Y轴) 和该基因在被检测到的细胞中的平均RNA计数 (X轴) 共同筛选后被排除的基因。图片标题中的基因数为筛选出的基因数,推荐控制在8k-16k
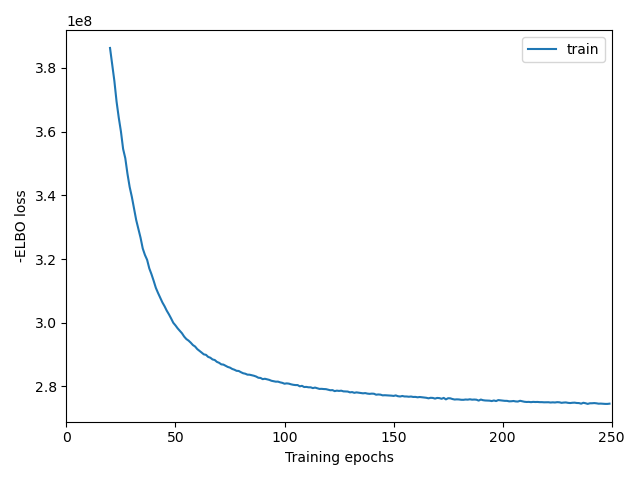
- 训练Loss下降图:
<reference_name>_train_history.png/pdf训练过程中ELBO loss的变化曲线,图中已去除前20个epoch的数据
- Cell2location构建的单细胞ref csv:
<reference_name>_inf_aver.csv每一行为一个基因,每一列为一个细胞类型,数值为cell2location计算的细胞类型特征 (用负二项回归模型计算的每个细胞类型每个基因的预估表达量)
| B_act | B_naive | CD4_CXCL13 | ... | |
|---|---|---|---|---|
| 7SK | 0.3071783 | 0.22791654 | 0.059129756 | ... |
| A1BG | 0.18173707 | 0.096046284 | 0.0936929 | ... |
| A1BG-AS1 | 0.04608244 | 0.042425267 | 0.08740552 | ... |
| A1CF | 0.00167472 | 0.000960604 | 0.002093679 | ... |
| ... | ... | ... | ... | ... |