Squidpy算法
用途
使用 Squidpy 工具包进行全面的空间组学分析,包括细胞交互模式(邻里富集)、共现性分析以及细胞类型的空间分布模式评估(Ripley's 统计)。
运行方式
SDAS spatialRelate squidpy -i st.h5ad -o outdir --label_key anno_cell2location \
--spatial_coords_scale 0.5 \
--bin_size 50.0 \
--n_neighs 9 \
--coord_type grid \
--n_perms_enrichment 1000 \
--cooccurrence_interval '100,1000,10' \
--n_simulations_ripley 100 \
--ripley_modes 'F,G,L' \
--n_cpus 24 \
--seed 42
输入参数说明
| Squidpy 参数 | 是否必须 | 默认值 | 描述 |
|---|---|---|---|
| -i / --input | 是 | 输入的 AnnData h5ad 文件路径。要求包含空间坐标和细胞注释。 | |
| -o / --output | 是 | 分析结果的输出文件夹路径。 | |
| --label_key | 是 | AnnData 对象中 (adata.obs) 用于细胞类型或区域聚类注释的列名。 | |
| --spatial_coords_scale | 否 | 0.5 | 应用于空间坐标的全局缩放因子。例如,对于stereo-seq,该值设为0.5,将坐标转为微米,方便结果解读。 |
| --min_cells_per_type | 否 | 20 | 为纳入分析,每种细胞类型/聚类所需的最少细胞(或 spot)数量。重要:建议设置为20及以上,过小的值可能导致分析不稳定。 |
| --n_cpus | 否 | 8 | 并行计算时可使用的 CPU核心数。CPU核数越多,运行时间相应较少,可根据计算资源合理设置。 |
| --bin_size | 否 | 50.0 | 原始数据坐标单位中 spot 的直径。此值将与 spatial_coords_scale 相乘用于确定 spatial_scatter 图中的最终绘图标记大小。 |
| --n_neighs | 否 | 9 | sq.gr.spatial_neighbors 中用于构建空间图的邻居数量。 |
| --coord_type | 否 | grid | 传递给 squidpy.gr.spatial_neighbors 的 coord_type 参数。可选值为:'generic' (适用于不规则排列的空间数据点,基于实际坐标计算邻接) 或 'grid' (默认,适用于排列在规则网格上的数据点,基于网格位置构建邻接,通常更快)。 |
| --n_perms_enrichment | 否 | 1000 | 邻域富集分析中用于置换检验的次数,以评估观察到的富集/耗竭的显著性。必须为正整数。 |
| --cooccurrence_interval | 否 | "100,1000,10" | 共现分析的距离区间定义,格式为 "起始距离,结束距离,点数"。例如 "50,250,10" 表示从距离50到250,均匀取10个距离点进行分析。起始距离必须非负,结束距离必须大于起始距离,点数必须为正整数。 |
| --n_simulations_ripley | 否 | 100 | 计算 Ripley's F, G, 或 L 函数时,用于生成置信区间的模拟次数。必须为正整数。 |
| --ripley_modes | 否 | "F,G,L" | 需要计算的 Ripley 统计模式,以逗号分隔 (例如 "F,G", "L")。支持的模式包括 "F", "G", "L"。 |
| --seed | 否 | 42 | 用于可复现性的随机种子。必须为非负整数。 |
输出结果展示
| 结果文件 | 描述 |
|---|---|
<input_name>_squidpy_spatial_scatter.pdf/png |
根据 --label_key 对细胞/spot 着色的空间分布图。 |
<input_name>_squidpy_nhood_enrichment_zscores.csv |
邻域富集分析的 Z-score 矩阵,表示细胞类型间的空间邻近关系强度。 |
<input_name>_squidpy_nhood_enrichment.pdf/png |
邻域富集分析 Z-score 矩阵的热图可视化。 |
<input_name>_squidpy_co_occurrence_scores_full.csv |
主要定量结果。该文件为长表格式,记录了每一对细胞类型在每个距离分箱下的原始共现得分,字段包括参考细胞类型、邻居细胞类型、距离、距离区间索引、共现得分。 |
<input_name>_squidpy_co_occurrence_all_types.pdf |
所有细胞类型对(或指定细胞类型)的共现概率随距离变化的趋势图 (多页 PDF)。 |
<input_name>_squidpy_co_occurrence_plots_png/ (文件夹) |
包含每个细胞类型与其它类型共现趋势的独立 PNG 图片。 |
<input_name>_squidpy_co_occurrence.pdf/png |
单个细胞类型的共现趋势图。 |
<input_name>_squidpy_ripley_<mode>_function.pdf/png |
Ripley's 统计指定模式 (F, G, 或 L) 的结果图,用于评估细胞类型的空间分布模式(聚集、分散或随机)。为每个在 --ripley_modes 中指定的模式生成一个文件。 |
<input_name>_squidpy_processed.h5ad |
经过 Squidpy 分析和预处理后,包含所有计算结果(通常存储在 adata.uns 中)的最终 AnnData 对象。 |
- 邻域富集分析Z-score矩阵:
<input_name>_squidpy_nhood_enrichment_zscores.csvSquidpy输出结果展示细胞类型间的空间邻近关系强度,Z-score值表示相对于随机分布的富集或耗竭程度
| B | Endo | Epi | Fib | Mye | NK | T | |
|---|---|---|---|---|---|---|---|
| B | 0.0 | -0.5 | -1.2 | 0.8 | 1.5 | 2.1 | -0.3 |
| Endo | -0.5 | 0.0 | 0.7 | -0.2 | -1.1 | 0.4 | 0.9 |
| Epi | -1.2 | 0.7 | 0.0 | 1.8 | -0.6 | -0.8 | 0.3 |
| Fib | 0.8 | -0.2 | 1.8 | 0.0 | 0.5 | -0.1 | 1.2 |
| Mye | 1.5 | -1.1 | -0.6 | 0.5 | 0.0 | 0.7 | -0.4 |
| NK | 2.1 | 0.4 | -0.8 | -0.1 | 0.7 | 0.0 | 1.6 |
| T | -0.3 | 0.9 | 0.3 | 1.2 | -0.4 | 1.6 | 0.0 |
- 共现性分析原始结果表:
<input_name>_squidpy_co_occurrence_scores_full.csv记录每一对细胞类型在每个距离分箱下的原始共现得分,适用于后续自定义分析和可视化。
| from_celltype | to_celltype | distance | distance_interval_index | cooccurrence_score |
|---|---|---|---|---|
| B | B | 100.0 | 0 | 0.5327037 |
| B | B | 200.0 | 1 | 0.5388954 |
| B | B | 300.0 | 2 | 0.47285548 |
| B | B | 400.0 | 3 | 0.44997987 |
| B | B | 500.0 | 4 | 0.41206497 |
| B | Endo | 100.0 | 0 | 0.12482233 |
| B | Endo | 200.0 | 1 | 0.1646443 |
| B | Endo | 300.0 | 2 | 0.15779616 |
| B | Endo | 400.0 | 3 | 0.20279443 |
| B | Epi | 100.0 | 0 | 0.06660331 |
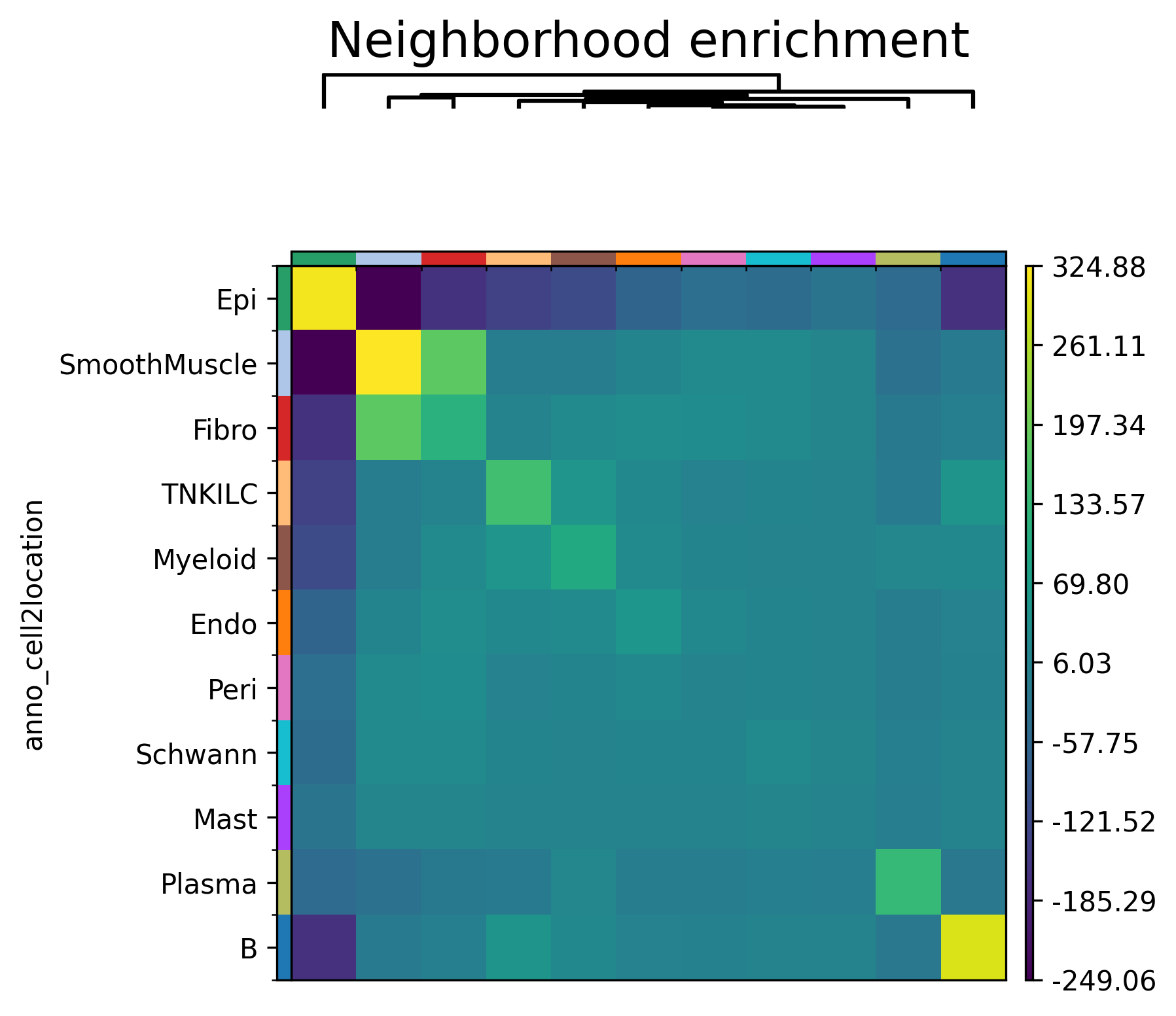
- 邻域富集分析热图:
<input_name>_squidpy_nhood_enrichment.png可视化邻域富集分析Z-score矩阵,图中颜色表示不同细胞类型对之间的空间邻近关系强度
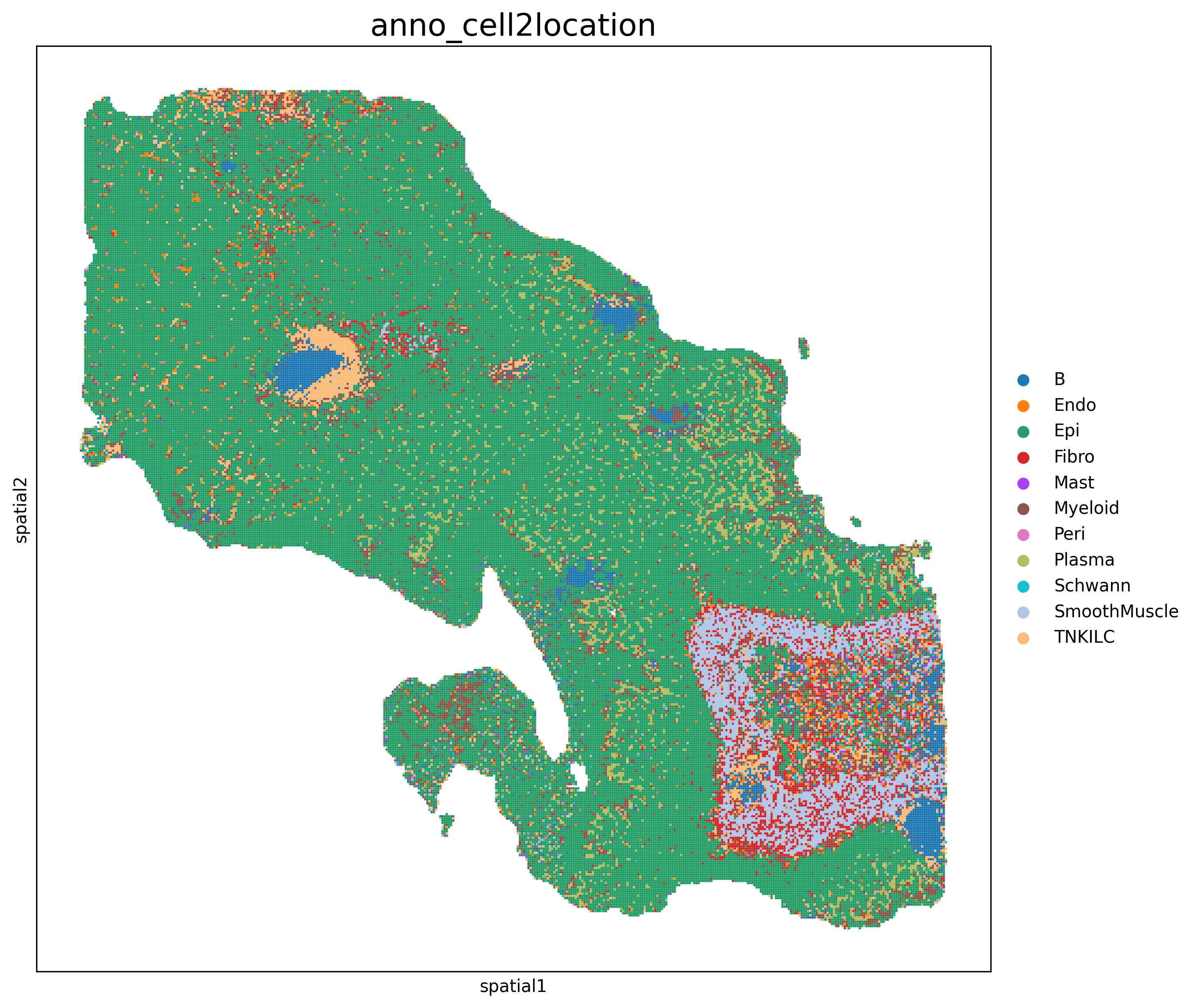
- 空间分布散点图:
<input_name>_squidpy_spatial_scatter.png根据细胞类型对细胞/spot着色的空间分布图,展示不同细胞类型在空间中的分布模式
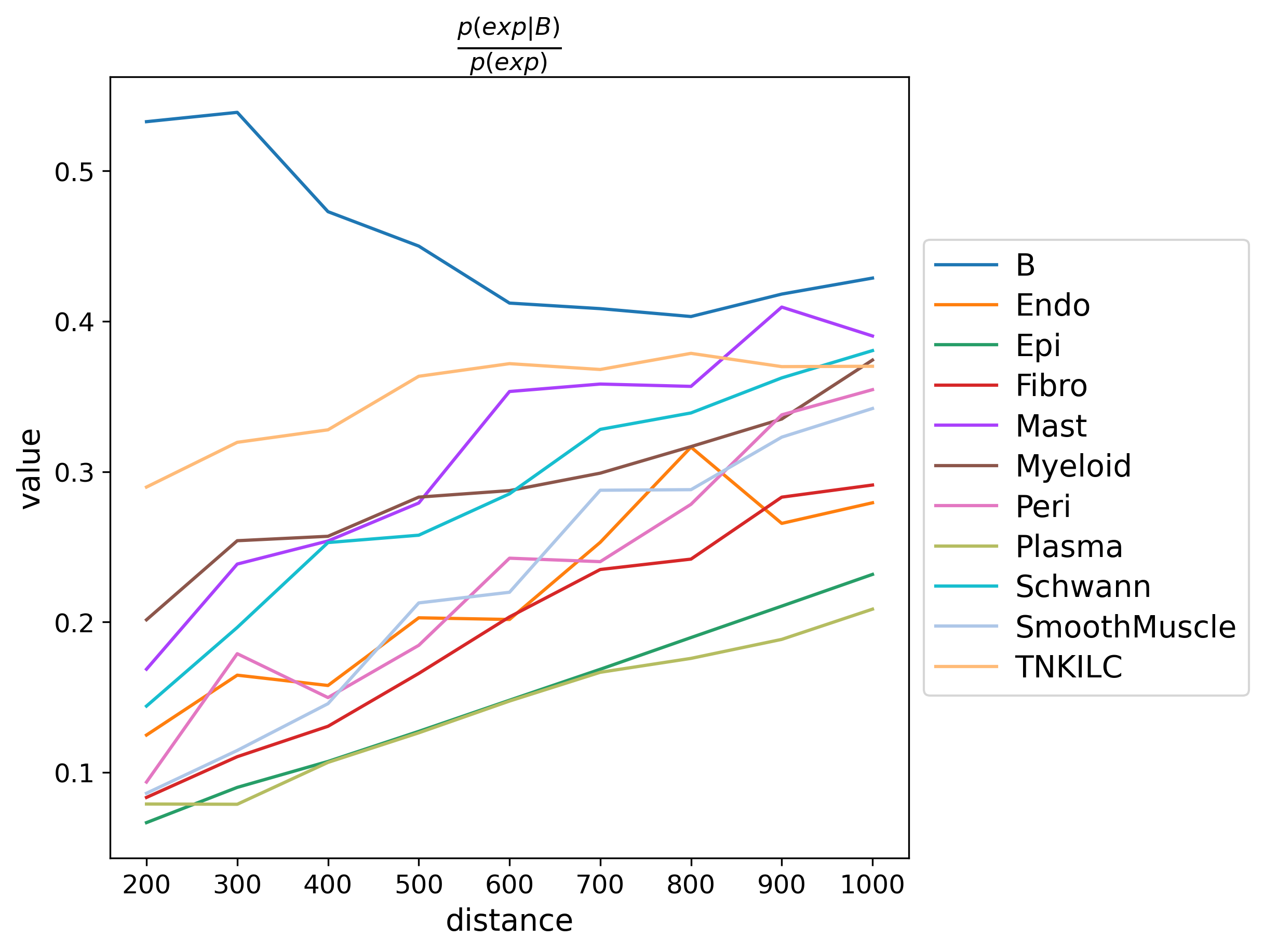
- 共现性分析趋势图:
<input_name>_squidpy_co_occurrence_B.png展示B细胞与其他细胞类型的共现概率随距离变化的趋势图,X轴为距离,Y轴为共现概率
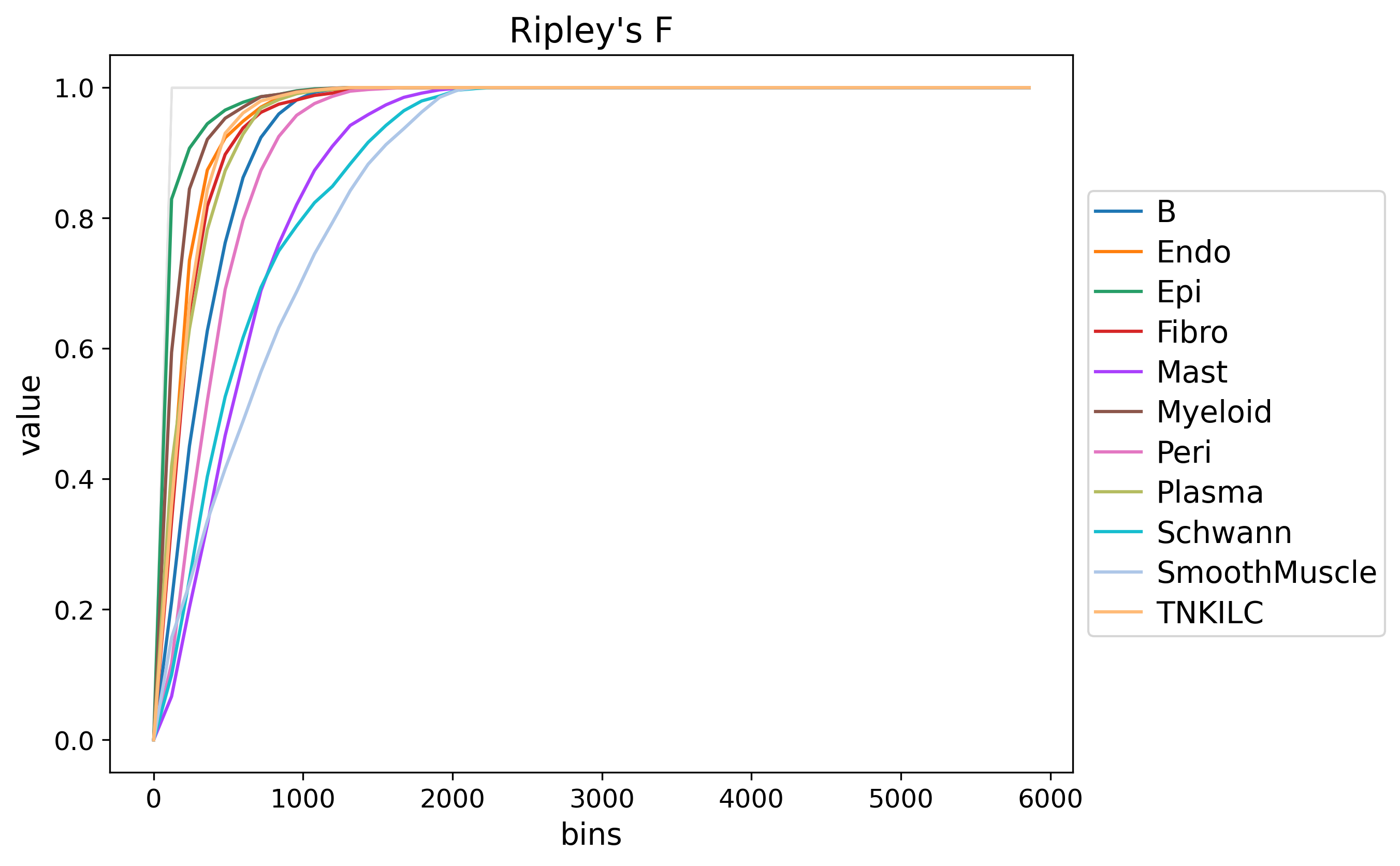
- Ripley's F函数分析:
<input_name>_squidpy_ripley_F_function.png展示细胞类型的空间分布模式,与随机分布的期望和置信区间进行比较
结果解读说明
- 邻域富集分析Z-score:正值表示富集(细胞类型倾向于邻近),负值表示耗竭(细胞类型倾向于远离),绝对值越大表示关系越显著
- 共现性分析:展示细胞类型随距离变化的共现模式,有助于理解细胞类型间的空间相互作用
- Ripley's统计:评估细胞类型的空间分布模式,F/G/L函数值高于随机期望表示聚集分布,低于随机期望表示分散分布
调参建议
- 若样本细胞类型较少或数据稀疏,建议调整
n_perms_enrichment和n_simulations_ripley参数以获得更稳定的结果 n_perms_enrichment:邻域富集分析的置换次数,建议根据数据规模调整,数据量大时可适当减少以节省计算时间n_simulations_ripley:Ripley's统计的模拟次数,影响置信区间的准确性,可根据需要调整cooccurrence_interval:共现分析的距离区间,建议根据细胞类型的大小和空间分布特征调整bin_size:影响空间散点图的点大小,可根据数据密度和可视化需求调整