cellchat算法
用途
使用cellchat进行细胞通讯分析
运行方式
SDAS CCI cellchat -i st.h5ad --label_key anno_spotlight -o outdir \
--add_spatial \
--bin_size 100 \
--species human \
--type truncatedMean
输入参数说明
| 参数 | 是否必须 | 默认值 | 描述 |
|---|---|---|---|
| -i / --input | 是 | h5ad文件,要求有原始表达矩阵 | |
| -o / --output | 是 | 输出文件夹 | |
| --label_key | 是 | 细胞类群的列名 | |
| --layer | 否 | h5ad存有原始表达矩阵的layer名称 | |
| --gene_symbol_key | 否 | real_gene_name | h5ad文件中表示基因名(symbol)的列名(_index 表示用h5ad.var.index) |
| --add_spatial | 否 | 如果分析时需考虑空间信息,则使用”--add_spatial” | |
| --bin_size | 否 | 如果使用“--add_spatial”,则需要提供bin_size, 如cellbin,10, 20, 50,100等 | |
| --species | 否 | human | cellchat内置的数据库,支持human、mouse、zebrafish,默认‘human’;当指定--cellchat_database时,该参数不起作用 |
| --cellchat_database | 否 | 自定义的cellchat数据库文件路径 | |
| --type | 否 | triMean | 计算每个细胞类型的基因表达平均值的方法,triMean或truncatedMean |
| --trim | 否 | 0.1 | type为truncatedMean时,可调整trim,trim越小找到的交互越多,trim为0.1时表示去掉两端各10%的极端值后再计算均值 |
| --spatial_coords_scale | 否 | 0.5 | 应用于空间坐标的全局缩放因子。例如,对于stereo-seq,该值设为0.5,将坐标转为微米,方便结果解读 |
| --scale_distance | 否 | 2 | 空间距离的归一化因子,比较不同 CellChat 对象之间的通信时,应使用相同的缩放因子。对于单个样本分析,不同的归一化因子不会影响配受体信号的排序 |
| --pathway.file | 否 | 自定义通路文件,用于生成_pathway_communication_network.png/pdf,png为每个pathway一张png,pdf是所有pathway绘制在一个pdf文件,使用“--add_spatial”时,输出空间网络图 | |
| --seed | 否 | 42 | 随机种子 |
自定义构建数据库可参考cellchat官网tutorial:
输出结果展示
| 结果文件 | 描述 |
|---|---|
<input_name>_cellchat_LR.csv |
互作的配受体结果(没有找到显著配受体时不输出) |
<input_name>_cellchat_LR_pathway.csv |
配受体富集的通路结果(没有找到显著配受体时不输出) |
|
展示各细胞类群间配受体互作强度的circle图和heatmap图,同时输出pdf和png |
|
展示各细胞类群间配受体互作数量的circle图和heatmap图,同时输出pdf和png |
<input_name>_signalingRole_scatter.png/pdf |
展示各细胞类群在信号网络中的角色分布,同时输出pdf和png |
<input_name>_pathway_communication_network.png/pdf |
指定通路的细胞间网络图,使用“--add_spatial”时,则输出空间网络图,同时输出pdf和png(如果指定通路没有显著富集则不输出) |
<input_name>_cellchat.rds |
包含细胞通讯结果的rds文件 |
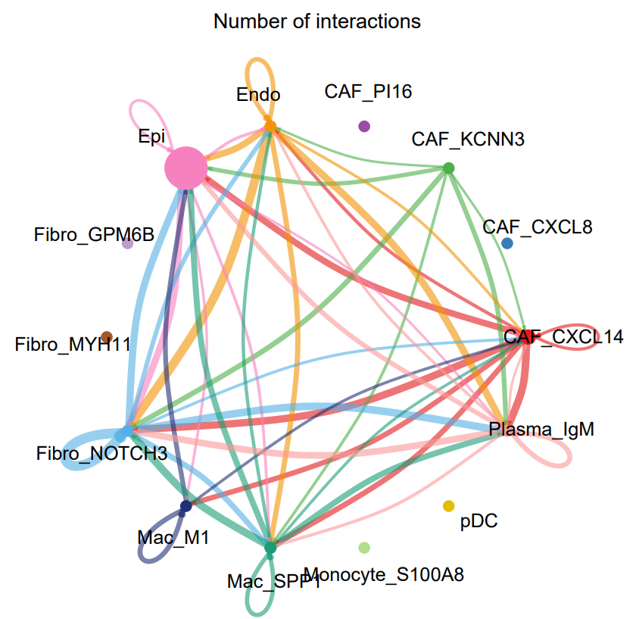
- 各细胞类群间配受体互作数量circle图:
<input_name>_number_of_interactions_circle.png/pdf展示各细胞类群间配受体互作数量,反映互作关系强弱。 圆圈表示细胞类群,连线表示配受体互作,连线粗细代表互作数量,圆圈大小代表该细胞类型参与的总相互作用数量,可直观比较哪些细胞类群之间互作最频繁
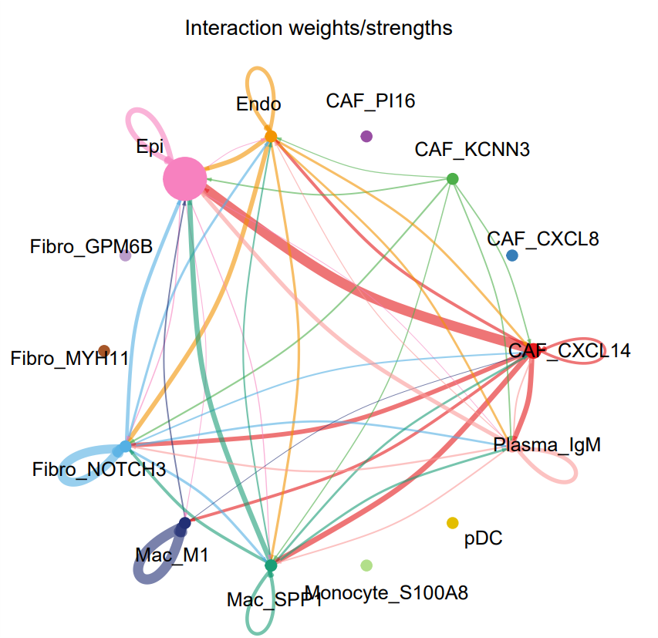
- 各细胞类群间配受体互作强度circle图:
<input_name>_interaction_strength_circle.png/pdf展示各细胞类群间配受体互作强度,反映信号传递活跃程度。 圆圈表示细胞类群,连线表示配受体互作,连线粗细代表互作强度,圆圈大小代表该细胞类型参与的总相互作用强度,可直观比较哪些细胞类群之间信号传递最活跃
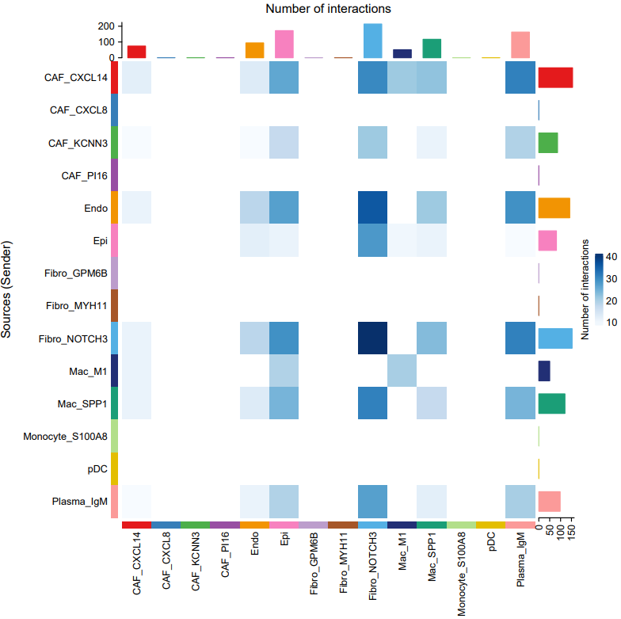
- 各细胞类群间配受体互作数量热图:
<input_name>_number_of_interactions_heatmap.png/pdf以热图形式展示互作数量,便于整体比较。 横轴和纵轴均为细胞类群,颜色深浅表示互作数量,颜色越深表示互作越多
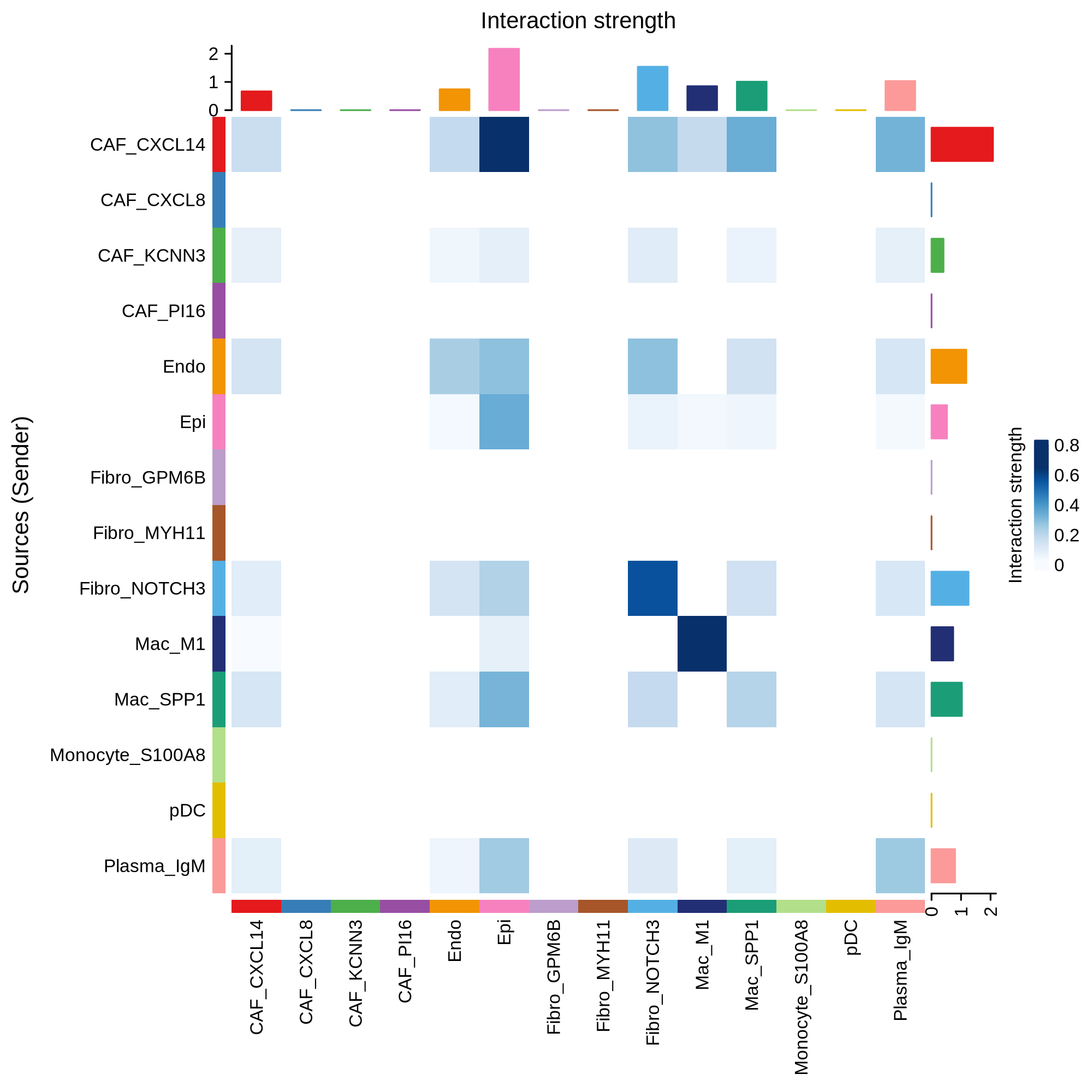
- 各细胞类群间配受体互作强度热图:
<input_name>_interaction_strength_heatmap.png/pdf以热图形式展示互作强度,便于整体比较。 横轴和纵轴均为细胞类群,颜色深浅表示互作强度,颜色越深表示信号传递越强
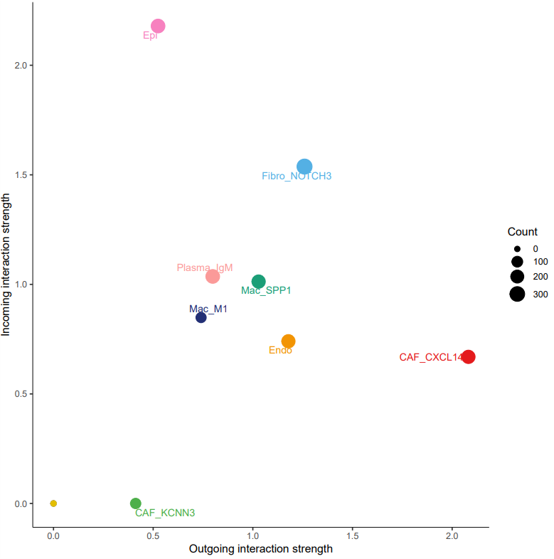
- 信号网络角色分布散点图:
<input_name>_signalingRole_scatter.png/pdf展示各细胞类群在信号网络中的角色分布。 横轴和纵轴为不同的信号网络特征(如发送者、接收者、调节者等),每个点代表一个细胞类群,点的位置反映其在网络中的功能角色,可识别主要信号发送者和接收者
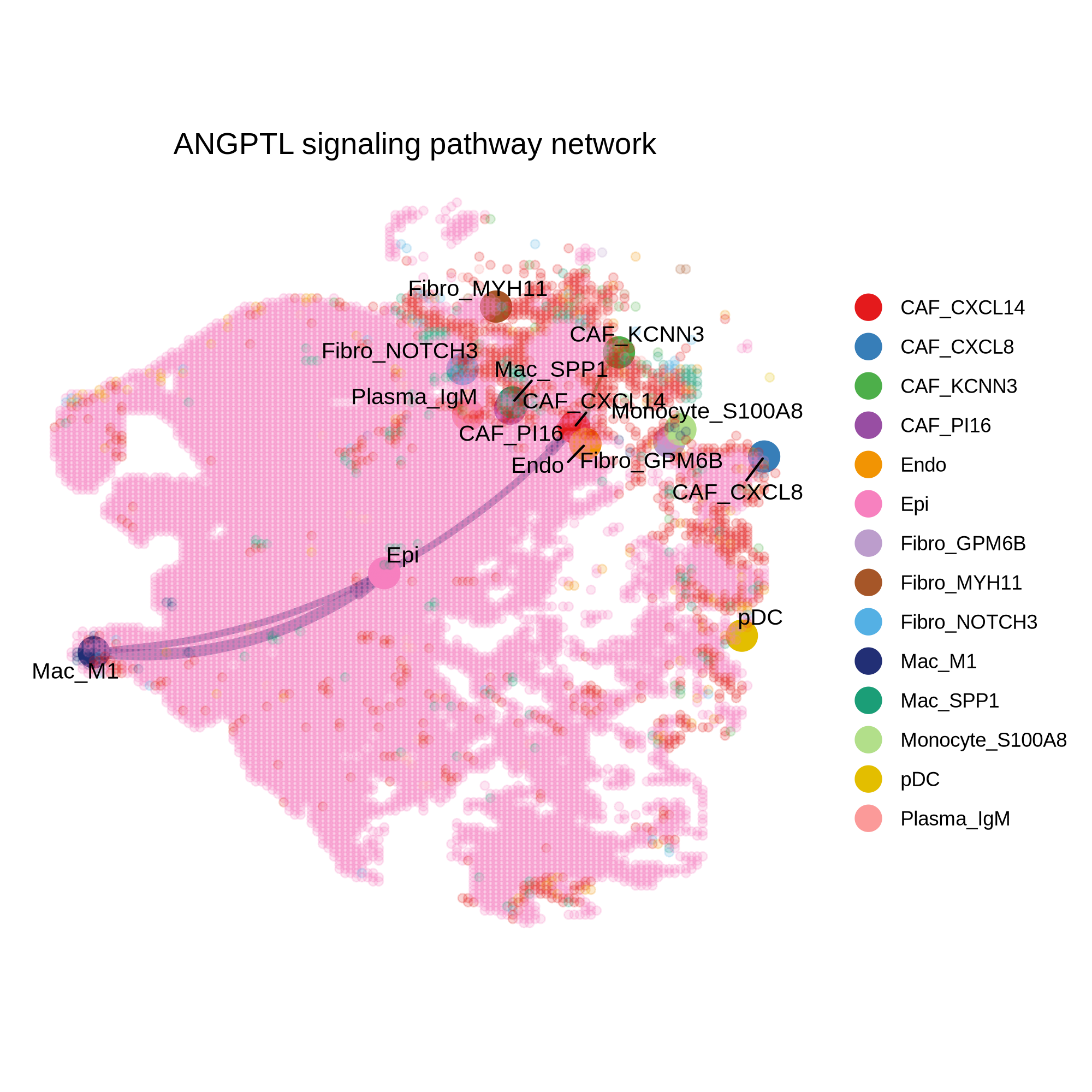
- 指定通路的空间网络图:
<input_name>_pathway_communication_network.png/pdf指定通路的细胞间网络结构,如果加空间信息分析(--add_spatial),则输出指定通路的空间网络图。 横纵坐标为空间坐标,每个点为细胞类群,连线表示通路相关的细胞通讯,连线粗细表示互作强度,节点圆圈大小代表该细胞类型参与的信号相互作用总数,可直观观察特定通路在空间中的分布和作用区域
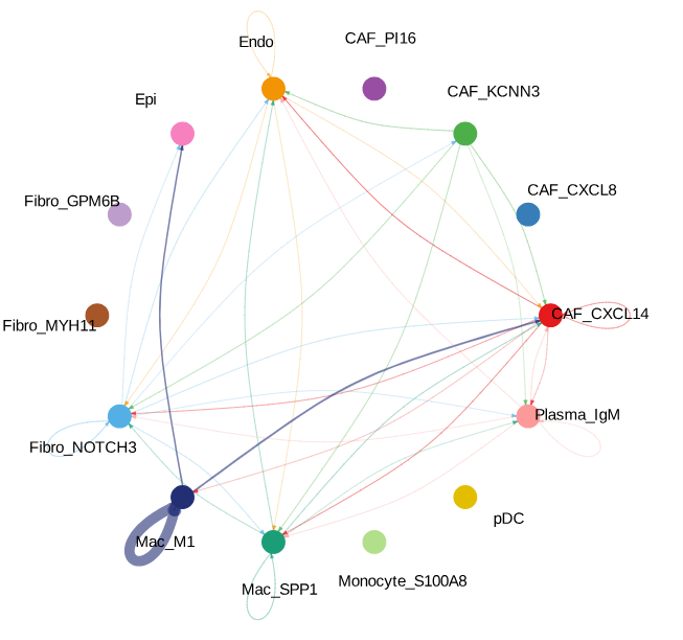
- 指定通路的细胞间网络结构图:
<input_name>_pathway_communication_network_nospatial.png/pdf指定通路的细胞间网络结构。 节点为细胞类群,连线表示通路相关的细胞通讯,连线粗细表示互作强度,节点圆圈大小代表该细胞类型参与的信号相互作用总数,适合无空间信息时观察通路网络结构
调参建议
type参数:推荐使用默认triMean,预测较强互作。如果希望获得更多的交互,可用 truncatedMean 并调整 trim (trim越小交互越多),trim为0.1时表示去掉两端各10%的极端值后再计算均值scale_distance:空间距离的归一化因子,比较不同 CellChat 对象之间的通信时,应使用相同的缩放因子。对于单个样本分析,不同的归一化因子不会影响配受体信号的排序