生存分析模块
用途
本模块基于IOBR、survival、survminer等R包,对免疫浸润/基因集打分结果与生存信息进行单因素生存分析,并输出标准化的生存曲线图。
输入文件示例
input特征打分/免疫浸润结果文件:每行一个样本名,每列为各类免疫细胞/基因集打分等特征,tab分割
| SampleID |
Macrophages_M2_CIBERSORT |
CD8_T_Cells_EPIC |
| Sample1 |
0.123 |
0.456 |
| Sample2 |
0.234 |
0.567 |
| Sample3 |
0.345 |
0.678 |
clinical生存信息文件:每行一个样本名,每列为生存时间、生存状态等特征,tab分割
| SampleID |
OS.time |
OS |
DFS.time |
DFS |
| Sample1 |
1000 |
1 |
800 |
0 |
| Sample2 |
800 |
0 |
600 |
0 |
| Sample3 |
1200 |
1 |
1000 |
1 |
运行方式
SDAS bulkValidate survivalKM --input tme_combine.txt --clinical survival.txt --signature Macrophages_M2_CIBERSORT --project_name survival --time OS.time --status OS.status --time_type day --output survival_output
输入参数说明
| 参数 |
必需 |
默认值 |
描述 |
| --input |
是 |
|
免疫浸润/打分结果文件路径 |
| --clinical |
是 |
|
生存信息文件路径 |
| --signature |
是 |
|
用于生存分析的特征列名 |
| --output |
是 |
|
输出目录路径 |
| --project_name |
否 |
test |
项目名称(用于输出文件命名等) |
| --time |
否 |
OS.time |
生存时间列名 |
| --status |
否 |
OS.status |
生存状态列名(0=生存/无复发,1=死亡/复发) |
| --time_type |
否 |
day |
时间单位,默认day |
输出结果展示
| 结果文件 |
描述 |
survival.png/pdf |
生存曲线图 |
- 生存曲线图:
survival.png/pdf展示指定特征分组下的生存分析曲线,显示高低组生存差异。
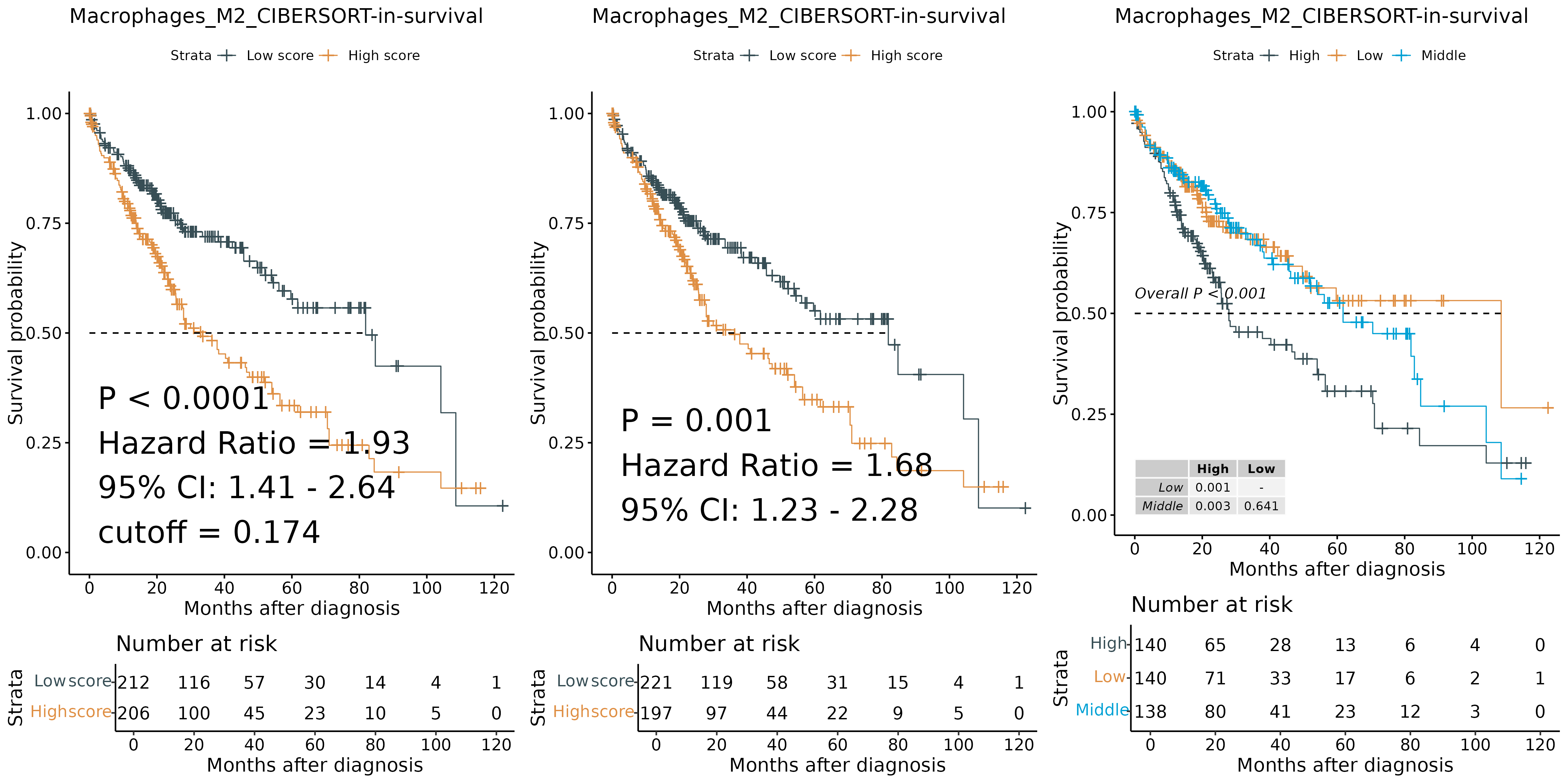
分组方法的区别:
- 最佳截断值(best cutoff)分组:通过统计方法寻找将两组生存差异最大化的截断值。通常能最显著地区分出高风险组和低风险组(如左图所示)。
- 平均数分组: 以所有样本得分的平均数作为分界点。这种方法在数据分布对称时效果较好,但在偏态分布时可能不够敏感(如中图所示)。
- 三等分组: 将样本按照得分分为三等分(低、中、高)。这种方法高低分组的差异可能不如前两种方法显著(如右图所示),但能避免极端值影响,并观察中间组的影响。
统计指标的含义:
- P值: 表示组间生存差异的显著性。P值越小,表明组间差异越不可能由随机因素导致。
- Hazard Ratio(风险比,HR): 表示高分组相对于低分组的死亡风险倍数。HR>1表示高风险
- 95% CI(置信区间): 表示HR的95%置信区间,反映估计的精确度。区间不包含1(如左图1.41-2.64)表示HR显著不等于1。区间越窄,估计越精确。
- Cutoff值:最佳截断值分组方法中使用的分界点。
- 生存曲线:展示了高分组(橙色)和低分组(蓝色)的生存概率随时间变化情况。曲线分离越明显,组间差异越大。
- 风险表(Number at risk): 在时间点下方显示各组的风险人数(即到该时间点还未发生终点事件的人数),这有助于评估随着时间的推移各组的样本数量变化。
