pyscenic算法
用途
本模块基于pyscenic分析细胞-基因表达矩阵,预测转录因子(TFs)与靶基因之间的相互作用
运行方式
SDAS TF --input test.h5ad --output TF_result --label_key anno_cell2location --slice_key group --celltype_list celltype.txt --species human --n_cpus 24
输入参数说明
| 参数 | 是否必须 | 默认值 | 描述 |
|---|---|---|---|
| -i/--input | 是 | 输入h5ad文件(可经过筛选后的) | |
| -o/--output | 是 | 输出文件夹,未创建则自动新建 | |
| --label_key | 是 | 指定h5ad.obs中细胞类型或分组的obs列名 | |
| --gene_symbol_key | 否 | real_gene_name | 指定h5ad中表示基因名(symbol)的列名 |
| --slice_key | 否 | 多h5ad.obs中分层分组的列名 | |
| --celltype_list | 否 | 需分析的细胞亚型列表文件路径。至少指定2种细胞类型,否则画图会报错。 | |
| --TF_list | 否 | 需关注的TF列表文件路径 | |
| --species | 否 | human | 物种,human/mouse |
| --db_names | 否 | motif/调控数据库文件路径,例hg38/10kbp*.feather | |
| --motif | 否 | motif注释文件路径,例hg38/motifs-*.tbl | |
| --n_cpus | 否 | 24 | 并行线程数 |
| --method | 否 | grnboost2 | GRN推断方法,grnboost2/genie3 |
输出结果展示
| 结果文件 | 描述 |
|---|---|
<input>_filtered.h5ad | (中间文件)使用目标celltype类型筛选后的h5ad文件 |
<input>.loom | (中间文件)基于h5ad构建的细胞-基因表达矩阵文件,pyscenic输入文件 |
adj.tsv | (中间文件)pyscenic构建的基因调控网络边文件,用于pyscenic ctx 构建TF-targets网络 |
sce_regulon.gmt | (中间文件)pyscenic构建的TF-targets网络,用于pyscenic aucell 计算Regulons(调控子)活性 |
sce_regulon_AUC.loom | pyscenic输出的regulon调控结果文件,每个细胞的活性得分, 用于后续可视化 |
Regulon_Heatmap.png/pdf | regulon活性热图,展示各细胞/分组的regulon活性 |
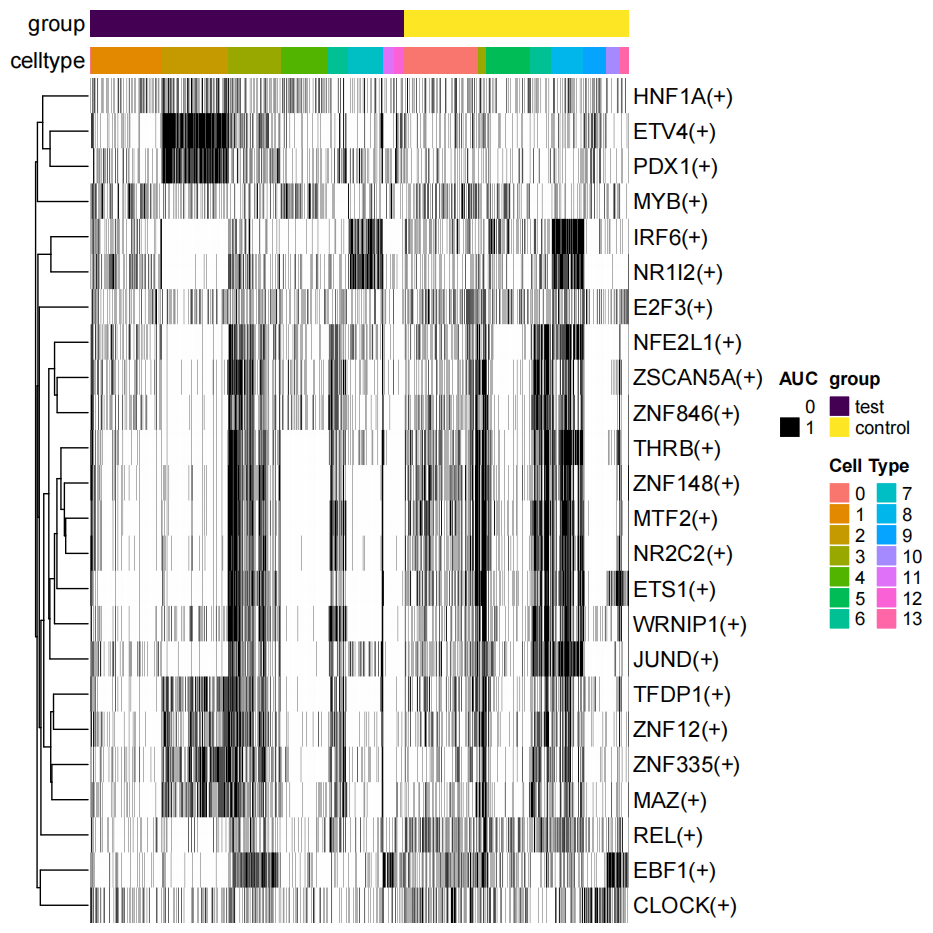
Binary_Regulon_Heatmap.png/pdf | regulon二值化活性热图,便于细胞亚群特异性分析 |
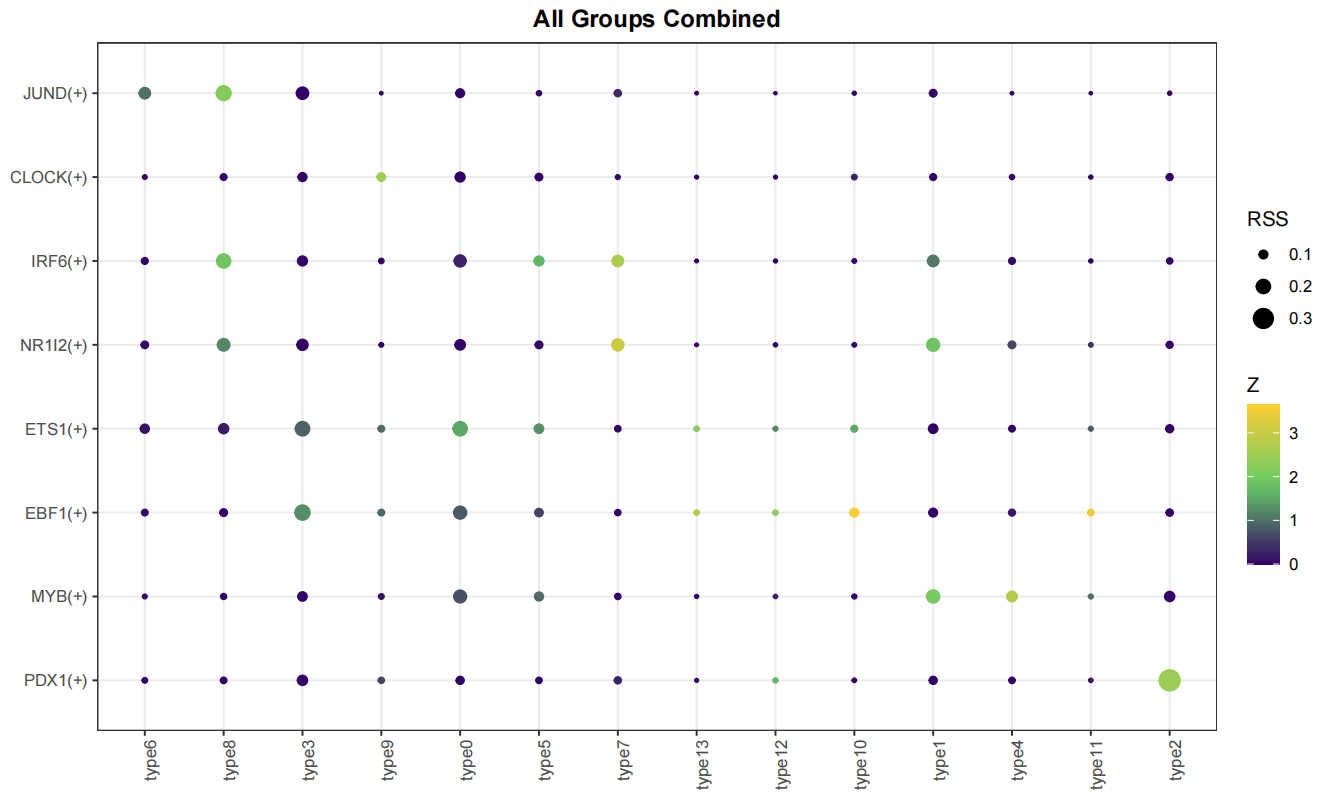
RSS.pdf/RSS_group*.png/pdf | regulon特异性得分RSS热图/分组热图 |
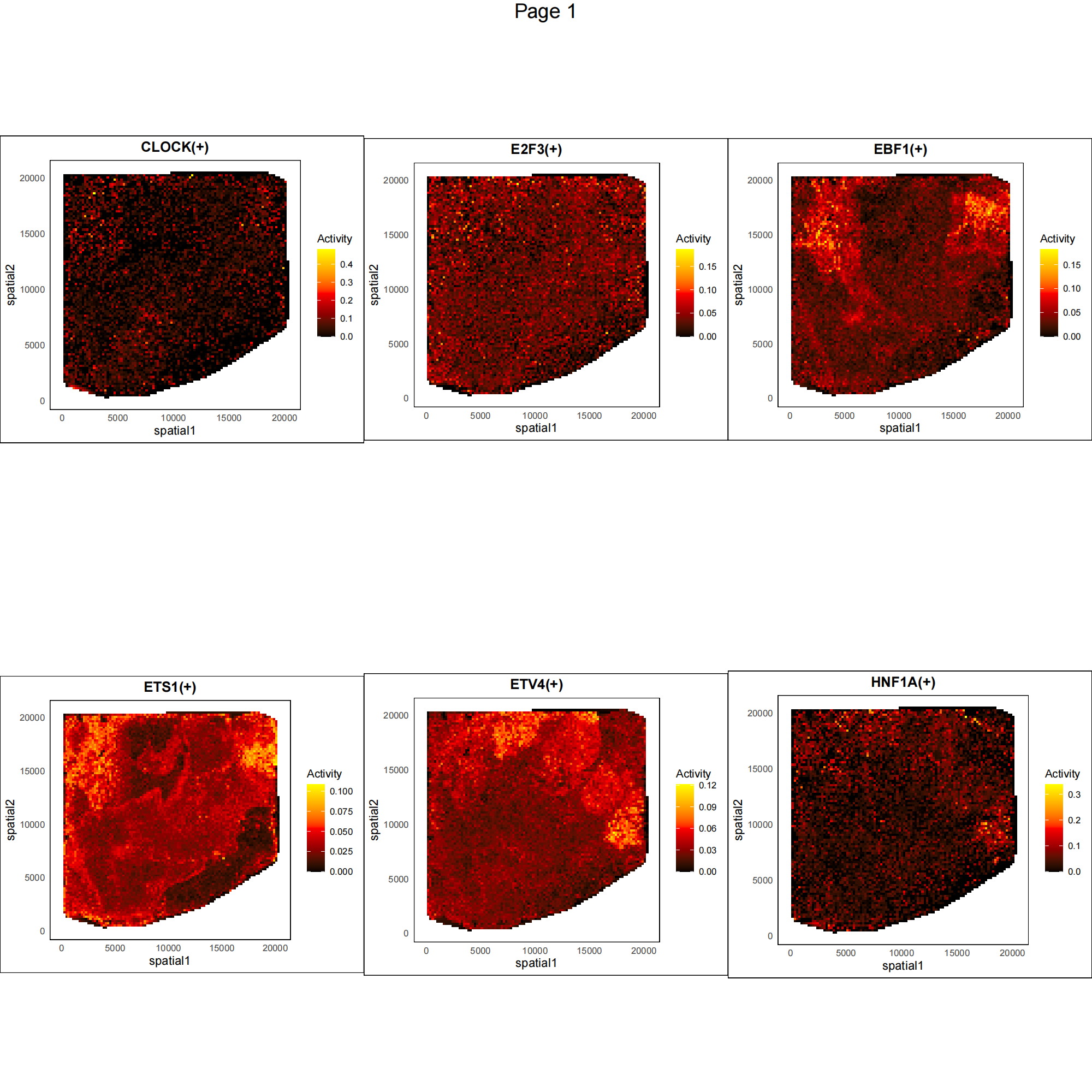
Spatial_Activity_Maps.png/pdf | regulon在空间上的活性热图,仅在--TF_list参数指定后绘制 |
- regulon活性热图:
Regulon_Heatmap.png/pdf每行一个regulon,每列一个细胞/分组,颜色表示AUC活性得分。用于比较同一Regulons在不同细胞中的活性,寻找Regulons特异性活化的细胞亚群等。
.png)
- regulon二值化活性热图:
Binary_Regulon_Heatmap.png/pdfregulon活性二值化后热图,黑色即该细胞开放该Regulons,便于细胞亚群特异性分析。
- regulon特异性得分RSS热图/分组热图:
RSS.png/pdfRSS_group*.png/pdfregulon在不同分组/细胞类型中的特异性得分RSS热图。圆圈大小表示RSS高低,颜色梯度表示标准化后的Z值大小;每行对应一个调控子,每列对应一种细胞类型。同一细胞类型内可直接比较调控子的特异性RSS,不同细胞类型可比较Z值。
- regulon空间活性热图:
Spatial_Activity_Maps.png/pdfregulon在空间上的活性分布,仅在空间转录组数据及输入--TF_list参数时输出。