基因集评分模块
用途
本模块基于AUCell、GSVA、IOBR等R包,对自定义基因集在bulk RNA-Seq数据的表达矩阵中的活性进行多种方法打分,并可视化分组热图
支持的基因集评分算法
- ssGSEA: 基于基因表达排序的累积分布计算富集分数,支持单样本分析
- GSVA: 核密度估计基因表达分布,输出标准化富集分数
- PCA: 基因表达标准化后主成分分析,取第一主成分得分
- Z-score: 计算基因集内基因的Z-score均值
- AUCell: 基于基因表达排名,计算基因集在前5%高表达基因的曲线下面积(AUC)
输入文件示例
expression表达矩阵文件:每行一个基因名,每列一个样本名,数值为对应的表达量,tab分割
| GeneID |
Sample1 |
Sample2 |
Sample3 |
| GENE1 |
1.234 |
2.345 |
3.456 |
| GENE2 |
4.567 |
5.678 |
6.789 |
clinical临床信息文件:每行一个样本名,每列一个临床特征,tab分割
| SampleID |
tissue_type.samples |
age |
gender |
| Sample1 |
Tumor |
45 |
Male |
| Sample2 |
Normal |
50 |
Female |
| Sample3 |
Tumor |
55 |
Male |
gene_set基因集合文件:表头为基因集合名称,每行一个基因名
| test_geneset |
|---|
| CD8A |
| CD8B |
| GZMA |
| GZMB |
运行方式
SDAS bulkValidate geneSetScore --expression fpkm.txt --gene_set geneset.txt --clinical clinical.txt --group_col tissue_type.samples --group_type discrete --output result_dir
输入参数说明
| 参数 |
是否必需 |
默认值 |
描述 |
| --expression |
是 |
|
表达矩阵文件路径。制表符分隔,行:基因ID,列:样本ID,值为FPKM/TPM等,不可为原始counts,不可log |
| --clinical |
是 |
|
临床信息文件路径。制表符分隔,行:样本ID,列:临床特征 |
| --group_col |
是 |
|
分组列名(需在临床信息文件中存在) |
| --gene_set |
是 |
|
自定义基因集文件路径。第一行为基因集名称(如test_geneset)后续每行一个基因名 |
| --output |
是 |
|
输出目录路径 |
| --group_type |
否 |
discrete |
分组类型:discrete/continuous,默认discrete |
输出结果展示
| 结果文件 |
描述 |
genescore_combine.txt |
所有打分方法的合并结果 |
geneset_score_heatmap.png/pdf |
不同打分方法分组热图 |
- 基因集评分结果表:
genescore_combine.txt 每行一个样本,每列为不同打分方法的结果。
| SampleID |
test_geneset_AUCell |
test_geneset_GSVA |
test_geneset_zscore |
test_geneset_ssGSEA |
test_geneset_PCA |
| Sample1 |
0.123 |
0.456 |
9.065 |
9.065 |
9.065 |
| Sample2 |
0.234 |
0.567 |
0.0677 |
0.0677 |
0.0677 |
- 基因集打分热图:
geneset_score_heatmap.png/pdf 展示所有样本指定基因集的表达分数。
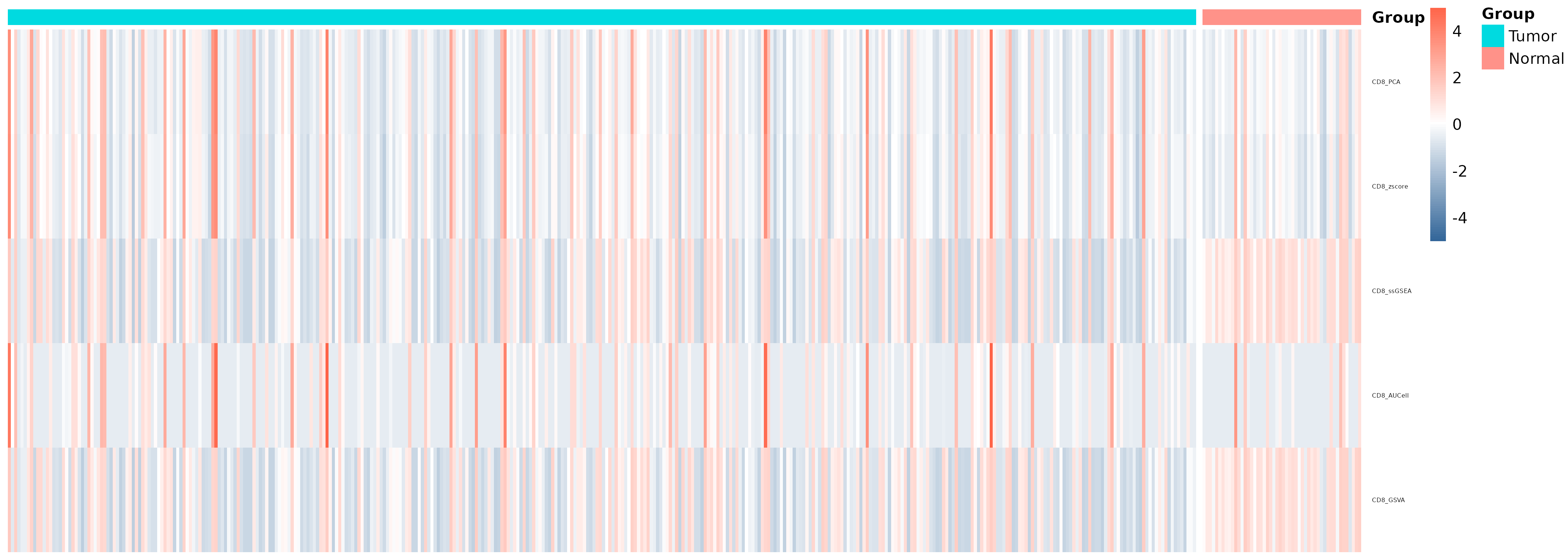
结果解读说明
- 基因集评分结果表:
genescore_combine.txt
- 若使用TCGA bulk转录组,优先选择GSVA或ssGSEA进行解读,这两种方法结果稳健,可支持复杂通路
- 若需保留基因相关性,优先选择PCA进行解读
- 若想快速分析,优先选择Z-score进行解读
