GSVA算法
用途与运行方式
场景一:指定具体对象进行gsva分析,例如指定leiden聚类中的1、2、3这几个cluster进行gsva分析
SDAS geneSetEnrichment gsva-i st.h5ad -o outdir \ --group_key leiden --idents 1,2,3 --species human场景二:对obs的某列做子集后再进行gsva分析,例如只对某种细胞类型的不同样本进行gsva分析
SDAS geneSetEnrichment gsva-i st.h5ad -o outdir \ --group_key leiden --idents 1,2,3 --species human \ --subset_key cell_type --subset_values B场景三:只用感兴趣的数据库进行分析
SDAS geneSetEnrichment gsva-i st.h5ad -o outdir \ --group_key leiden --idents 1,2,3 \ --gmt sdas_deg_enrichment/lib/GSEADB/h.all.v2024.1.Hs.symbols.gmt,sdas_deg_enrichment/lib/GSEADB/KEGG_2021_Human.gmt场景四:对obs中某列的所有元素一起进行gsva分析,要将某列中的所有元素放到一起进行gsva分析,此时
--idents参数应该写为特定字符allSDAS geneSetEnrichment gsva-i st.h5ad -o outdir \ --group_key leiden --idents all --species human \
输入参数说明
| gsva参数 | 是否必须 | 默认值 | 描述 |
|---|---|---|---|
| -i / --input | 是 | Stereo-seq h5ad,要求原始矩阵 | |
| --group_key | 是 | 需要进行gsva分析的对象所在的obs名称 | |
| --idents | 是 | 需要用于进行gsva分析的对象,用','分隔多个对象 | |
| -o / --output | 是 | 结果存放路径 | |
| --subset_key | 否 | 需要提取的信息所属的obs名称 | |
| --subset_values | 否 | 需要提取的信息,存在多个时用','分隔 | |
| --layer | 否 | 指定表达矩阵,不指定时使用adata.raw.X或adata.X | |
| --gene_symbol_key | 否 | real_gene_name | 指定gene name所在列,默认real_gene_name,如果没有则使用矩阵中的原始基因信息 |
| --species | 否 | human | 指定数据库,'human' 或 'mouse',默认 'human',当指定--gmt参数时,该参数不起作用 |
| --sample_size | 否 | 0 | 对输入文件的bin数量进行随机取样以减小内存消耗,默认值为0,不做采样 |
| --gmt | 否 | gmt格式的数据库文件,其中gene name信息必须为大写,多个文件时用','隔开 | |
| --kernel_cdf | 否 | Gaussian | 基于原始表达矩阵时选'Poisson',其他选'Gaussian' |
| -v / --verbose | 否 | 启用详细模式,打印任务进度。默认:False | |
| --mx_diff | 否 | 设置时,富集分数(ES)将计算为随机游走的最大偏离距离。默认:False | |
| --abs_ranking | 否 | 仅当未设置--mx_diff时生效,使用原始的Kuiper统计量计算方法。默认:False | |
| --min_size | 否 | 15 | 基因集中包含的输入基因最小数量。默认:15 |
| --max_size | 否 | 20000 | 基因集中包含的输入基因最大数量。默认:20000 |
| --weight | 否 | 1 | 定义GSVA随机游走中的τ(tau)参数。默认:1 |
| --seed | 否 | 123 | 随机数种子值。默认:123 |
| --threads | 否 | 1 | 并行计算使用的进程数。默认:1 |
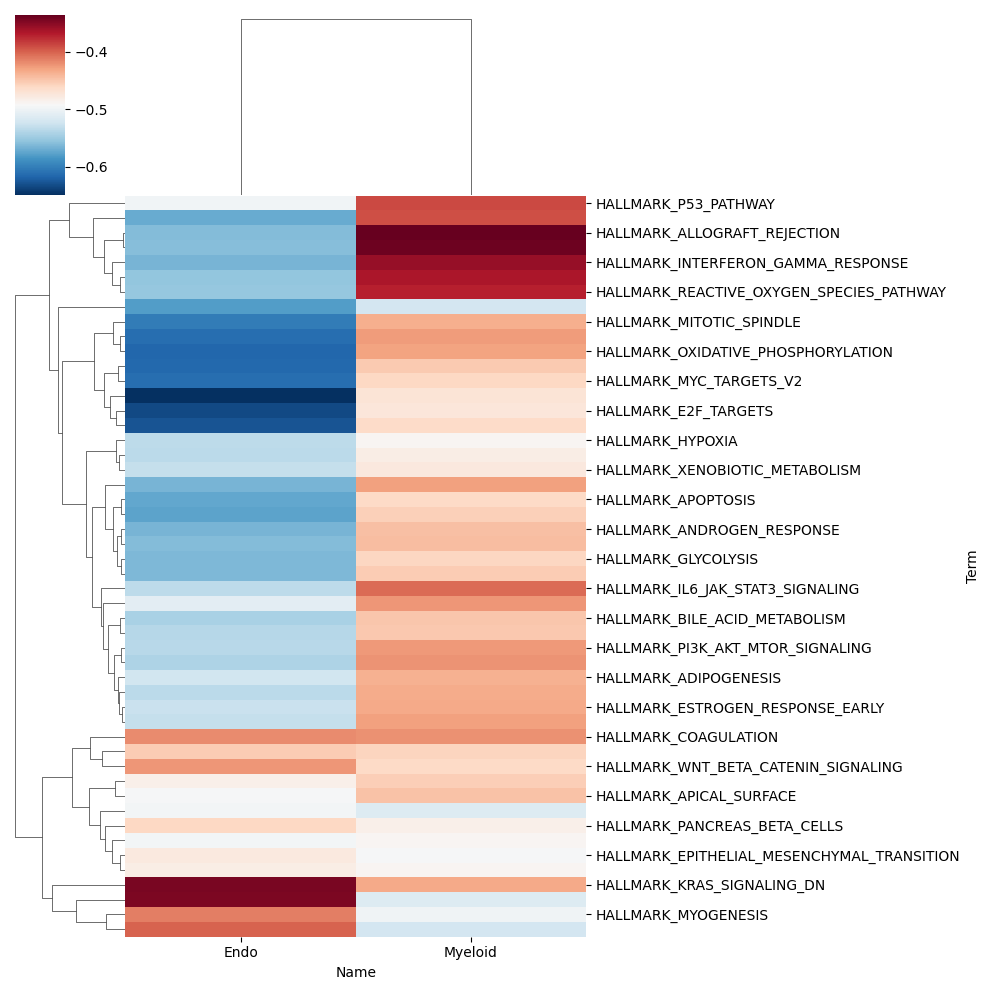
输出结果展示
| gsva结果文件 | 描述 |
|---|---|
GSVA.{database}.csv |
csv格式的结果文件 |
GSVA.{database}.pdf/png |
pdf和png格式的图像文件 |
- GSVA csv文件格式:
GSVA.{database}.csv,这个文件是gsva分析结果文件,第一列是Term表示功能名称,后面每一列表示一个样本,数值为正表示该样本在对应的功能上活性较高,数值为负表示活性较低。
| Term | ident1 | ident2 | ... |
|---|---|---|---|
| HALLMARK_ADIPOGENESIS | -0.32809425650271146 | -0.306805475112318 | .... |
| HALLMARK_ALLOGRAFT_REJECTION | -0.3052190348950549 | 0.22055475913564931 | .... |
| HALLMARK_ANDROGEN_RESPONSE | -0.39290236695613107 | -0.3080397441881526 | .... |
| ... | ... | ... | ... |
- gsva分析结果热图:
GSVA.{database}.pdf/png,图中纵列表示功能通路名称横列表示样本名称,图例表示gsva计算的得分。